오늘은 single-cell multiomics analysis를 위한 기술 중 single-cell RNA sequencing (scRNA-seq) methods를 정리해보겠습니다.
어제는 single cell whole genomcing sequencing methods인 MDA와 MALBAC에 대해 정리했었죠?
[대학원 준비 2일차] multiple displacement amplification(MDA) & multiple annealing and looping-based amplification cycles
이번엔 single-cell multiomics analysis를 위한 시퀀싱 기술들을 알아보겠습니다. 우선 single-cell sequencing 기술들은 아래 유튜브에서 잘 설명해주었으니 영상을 보시길 추천드립니다. 아래 영상은
tkmstudy.tistory.com
우선, RNA sequencing이 중요한 이유는 몸의 설계도 정도인 DNA와 달리 RNA를 살펴보면, 몸 속 현장에서 수많은 유전자 중 어떤 유전자가 발현되어 사용되는지를 이해할 수 있기 때문입니다.
즉, 질병 프로세스나 신약 적용에 있어 어떤 유전자가 덜 사용되고 더 사용되는지를 이해함으로써 기존 치료법의 효능을 높이거나 새로운 신약후보물질을 발굴할 때 사용할 수 있습니다.
또한 개별 세포들 사이에서 유전자 발현의 차이를 이해함으로써 정상세포와 암세포, 당뇨병 환자와 정상 환자의 개별 세포 수준에서의 transcriptome을 비교하여 관련 치료전략을 세우는데에도 도움을 줄 수 있습니다.
위 영상에서 single-cell RNA sequencing에 대해 잘 설명해주고 있는데요,
우선 본 기술의 목적은 heterogenous한 tissue에서 individual cells의 gene expression profile을 분석하기 위함이라고 합니다.
그럼으로써 마치 화채에 어떤 과일이 들어있는지 파악하듯이, 조직의 어떤 세포에서 어떤 유전자가 발현되었는지를 이해할 수 있다고 합니다.

본 분석을 위해 cell isolation을 위해 tissue는 trypsin이나 papain에 의해 dissociated되고,
그렇게 single cell suspension이 만들어지고 나면, 배양 후 hemocytometer를 활용해 suspension에 얼마나 cells이 있는지 계산합니다.
아래 영상에서는 hemocytometer의 사용법과 trypan blue에 의한 dillution factor를 고려해 viable cells의 density(white : 살아있는 세포, dark blue : 죽은세포)를 계산하는 방법을 소개하고 있습니다.
이후엔 barcoded oligonucleotides가 포함된 GEM Beads를 single cells와 oil에 흘려보내는 장치를 통해
특정 세포에 특정 beads가 함께 emulsion 되도록 하여 single cell GEM emulsion을 생성합니다.
이러한 방식은 Droplet-based 방식으로 unique molecular identifiers(UMIs)와 cell barcodes로 cell and gene specific identification을 가능하게 하여
high cell throughput과 good data quality를 갖는다4)고 합니다.

절차적으로 Beads가 mRNA를 capture할 수 있는 형태라 cell을 lysis시키고 나면, Beads의 OligodT(TTTTT..)에 mRNA(AAAA..)가 결합되고,
아래 이미지처럼 mRNA에 대해 reverse transcription*으로 cDNA(complementart DNA)가 형성됩니다.
* reverse transcription : RNA를 template로 complementary DNA를 합성하는 과정, DNA로부터 RNA를 합성하는 central dogma를 역행하기에 역전사 과정
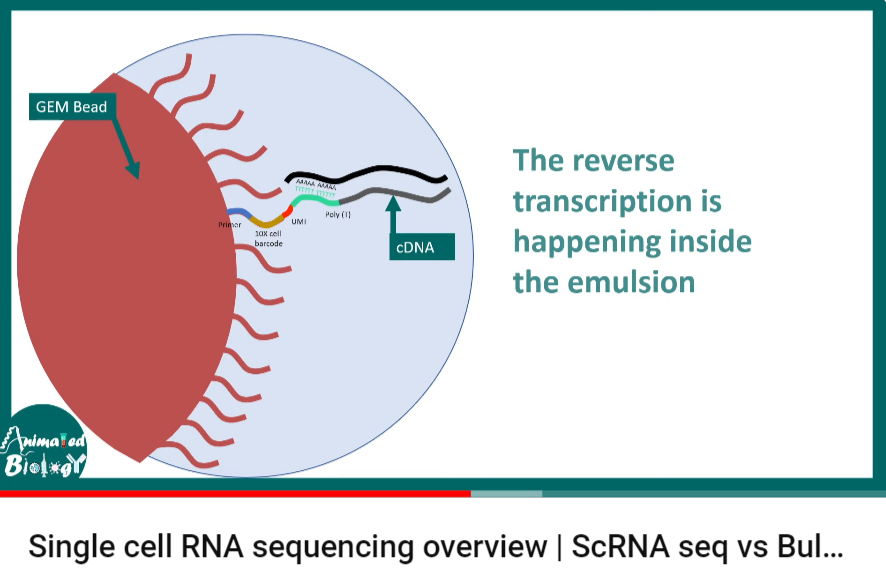
해당 cDNA fragment에 대해 Enzymatic Fragmentation 등을 거쳐 library preparation을 해주고,
약 500bp를 갖는 library만 남기도록 quality control(QC)를 진행합니다. 그 다음에서야 sequencing이 진행이 됩니다.
참고로 single-cell sequencing은 상당한 양의 시간, 돈, 샘플 재료 등이 필요하기에 cell isolation, library preparation, sequencing 절차 전에 high-quality를 보장할 수 있는 quality control이 필수4)라고 합니다.
"The barcoded cDNA is then used to prepare a sequencing library, which includes the addition of sequencing adapters and PCR amplification. Finally, the library is sequenced with NGS." - 10Xgenomic3).
챗GPT에게 왜 시퀀싱 전에 sequencing adapters를 더하고, PCR amlification을 진행했는지 물어봤습니다.

번역하자면 sequencing adapters는 NGS platform에서 cDNA를 결합하고, 세포와 분자를 검증하기 위한 barcodes를 식별하는데 필수적이라고 하고,
PCR amlification single cells의 RNA의 양은 너무 적기에 sequencing material을 늘리기 위해 필요하다고 합니다.
위의 영상은 이전 영상에서 영상을 링크했던 Eric Chow란 분이 NGS sample preparation에 대해 소개해주는 영상인데,
10분 40초쯤 SMART-seq에 대해 소개해주는 거부터 해서 illumina의 sequencing adapters에 대해 소개하는 14분 30초까지 위의 library preperation 단계와 비슷한 것 같으니 참고바랍니다.
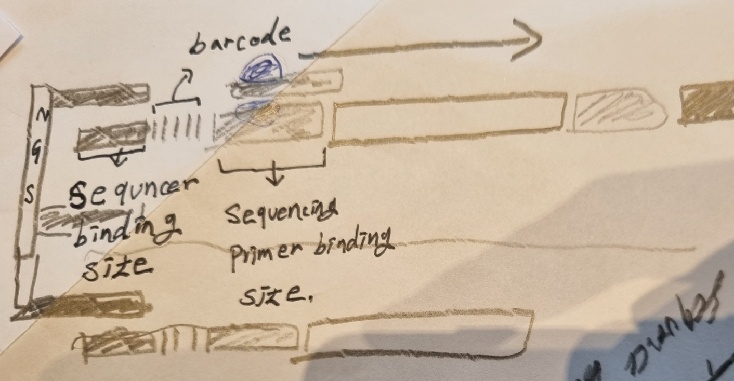
기초적으로 adapters의 구성(sequencer binding site, Index(barcode),sequencing primer binding site)에 대해선 아래 영상에서 잘 소개해주는 듯 하니 참고 바랍니다.
이후 reads를 reference에 대해 alignment를 시키는데 이때 소프트웨어 프로그램을 사용합니다.
아래는 HISAT를 활용해 RNA를 alignment하는 영상이니 참고바랍니다.
그러고 나면 transcriptome을 기반으로 identified된 개별 세포들에서 어떤 gene이 얼만큼 upregulated 혹은 downregulated되었는지 알 수 있습니다.
구체적으로 Cell과 Gene이 각각 열과 행으로 된 Single cell matrix를 도출할 수 있고, 본 Data를 시각화해서 보여주기도 하고
히트맵, bubble plot 등 downstream analysis를 통해 다각적으로 분석해볼 수도 있습니다.


결국 scRNA sequencing은 개별 세포의 유전자 발현을 분석함으로써 cellular heterogeneity
즉, cell-to-cell variation을 보다 specific한 관점에서 확인할 수 있도록 돕고
이를 통해 tumor cell과 normal cell의 genetic heterogeneity를 이해하는데 도움을 줄 수 있겠습니다.
마지막으로, ILLUMNIA에서 전반적인 single cell sequencing and analysis workflow를 정리한 영상을 링크하니 한번 보시길 추천드립니다.
또 다시 한번 정리하는 겸, DNALINK에서 정리한 single-cell RNA sequencing의 개념을 인용해보며 글을 마치려고 합니다.
Single Cell RNA Sequencing
단일 세포 전사체 분석(Single-cell RNA Sequencing)은 개별 세포 단위로 분리하여 RNA를 증폭하고 서열분석하여 해당 세포의 전사체적 특징을 분석하는 기술입니다. 대표적인 표준화된 분석 방법으로는
dnalink.com
단일 세포 전사체 분석(Single-cell RNA Sequencing)은 개별 세포 단위로 분리하여 RNA를 증폭하고 서열분석하여 해당 세포의 전사체적 특징을 분석하는 기술입니다. 대표적인 표준화된 분석 방법으로는 세포 단위로 구분 가능한 barcode를 RNA에 결합시켜 서열분석하는 것이며, 데이터 분석 과정에서 세포 수준의 연구 결과를 확보할 수 있습니다.. 이보다 더 발전된 기술로 Single Cell 단위의 Copy Number Variants, Immune profiling 및 Antibody를 이용한 Cell Surface Protein 분석도 가능하게 되었습니다. - 출처 : DNALINK
대표적인 표준화된 분석 방법으로는 세포 단위로 구분 가능한 barcode를 RNA에 결합시켜 서열분석하는 것이며, 데이터 분

마치기전에 single-cell RNA sequencing에 대해 brief view해주는 12분짜리 영상을 복습 겸 볼 수 있도록 첨부합니다.
영상 내용에서 보듯 해당 기술이 diabetes에서 downlation되는 gene과 upregulation되는 gene을 구분하는데에도 활용할 수 있는 듯 합니다.
다음 글에서는 scRNA sequencing methods 중 SMART-seq에 대해 자세히 알아보도록 하겠습니다.
참고자료
1) DNALINK, NGS > Single Cell Sequencing
2) Animated with Arpan, Single cell RNA sequencing overview | ScRNA seq vs Bulk seq | chemistry of ScRNA seq |Bio Techniques, Youtube
3) Natalya Ortolano, The neXt generation of single cell RNA-seq: An introduction to GEM-X technology. 10Xgenomics, 2024
4) IILUMINA, Illumina Single-Cell Sequencing Workflows: Critical Steps and Considerations, URL : https://www.illumina.com/ techniques/sequencing/rna-sequencing/ultra-low-input-single-cell-rna-seq.html



