이번엔 rRNA, tRNA에 이어 mRNA의 합성과 프로세스 과정에 대해 살펴보겠습니다.
[대학원 준비 5일차] 이론 복습 :: U20 snoRNA, U68 snoRNA, 그리고 tRNA(transfer RNA)
오늘은 4일 차 글에 이어 Transcription 관련 이론 복습을 해보도록 하겠습니다. [대학원 준비 4일차] 이론 복습 :: transcription in bacteria, rRNA preprocessing이번 글에서는 전 글에 이어 전공 수업 때 배운
tkmstudy.tistory.com
전에 bacteria에서 mRNAs가 합성되는 절차를 다뤘던 것 같은데, 이번엔 RNA polymerase II에 의해 진핵생물에서 mRNAs가 합성되는 절차를 설명해보겠습니다.
우선, RNA polymerase II는 general transcription factors (GTFs)의 도움으로 promoter에 결합하게 되며, 그렇게 preinitiation complex가 형성됩니다.
이때 중요한 promoter의 부분은 initaition site로부터 24~32 bases upstream에 놓여있고, 해당 영역은 oligonucleotide 5'-TATAAA-3'과 같거나 매우 유사한 consensus sequence를 포함합니다.
이를 TATA box라고 하는데 제가 prokaryotic cells의 pribnow box와 eukaryotic cells의 TATA box를 헷갈렸던 것 같습니다.
그리고 TATA-binding protein(TBP)은 promoter의 TATAbox를 인식하고,
promoter에 결합한 TBP는 DNA에서 conformational change를 야기하게 됩니다.
그 절차에 대해 시간은 걸리더라도 중요하니 기억에 남기기 위해 영상으로 만들어봤습니다.
차례대로 설명하자면, 우선 TBP unit과 또 다른 subunits인 TBP-associated factors(TAFs)를 포함한 TFIID가 TATA box로 갑니다.
이후 추가적으로 TFIIA와 TFIIB라는 GTF가 붙게 되고, 3가지 GTF(TBP, TFIIA, TFIIB)는 TFIIF와 함께하는 RNA polymerase의 binding을 유도하게 됩니다.
그리고 또 다른 GTF(TFIIE, TFIIH)가 complex에 붙으면서 polymerase를 활성화하게 되어 전사가 시작됩니다. 전사가 시작되는 절차는 이미지로 만들어봤습니다.

우선, TFIIH는 enzymatic activity를 가진 유일한 GTF로 알려져 있으며(Protien kinase가 RNA polymerase를 인산화도록 도움),
무엇보다 RNA polymerase II의 carboxyl-terminal domain (CTD)의 5번째 위치의 serin residues를 인산화(phosphorylates) 시킵니다.
그리고 TFIIH에 의한 인산화는 enzyme을 preinitiation complex로부터 벗어나도록 유발합니다.
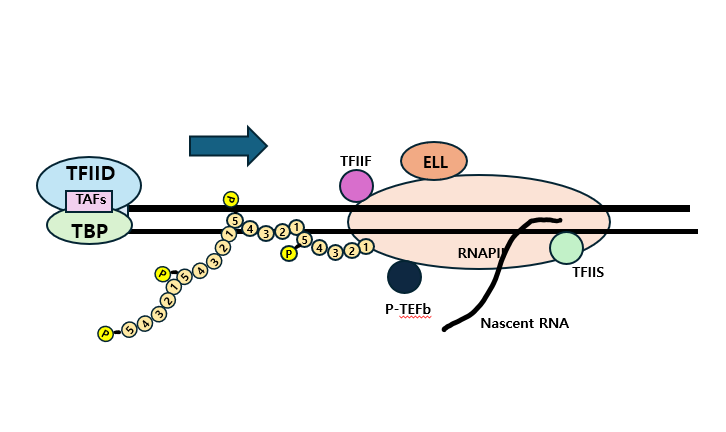
그렇게 RNAPII에 의해 mRNA transcription이 일어나는 과정에서 위 이미지에 보이는 P-TEFb는 두 번째 위치에 있는 serin residues를 인산화시키는데,
이는 RNA splicing과 poly(A) tail addition을 위한 추가적인 protein factors를 불러들입니다.
결국 transcription factors는 preintiation complex가 특정 promotor에 조립될 것인지 아닌지를 결정하는 것은 물론, polymerase가 promoter로부터 transciption을 개시하는 속도를 결정하게 됩니다.
앞서 설명한 진핵 세포에서 이루어지는 전사 과정에 대해 아래 영상에서 재밌는 비유와 함께 더 잘 소개해주고 있으니 참고 바랍니다(영상에 오류가 좀 있는 것 같긴 합니다).
그렇게 전사된 대부분의 mRNAs는 상당한 양의 noncoding segment를 가지는데
이러한 Noncoding 부분들은 mRNA의 5'과 3' end 모두에 발견되고, regulatory roles에 중요한 sequences를 포함합니다.
이후5' end는 methylated gaunosine cap (5' capping)를 갖고, 3' end는 adenosin residues로 된 poly A tail(3' tailing)를 갖게 되는데 그 과정을 소개하겠습니다.
먼저 mRNA의 구조는 다음과 같이 나타낼 수 있습니다.
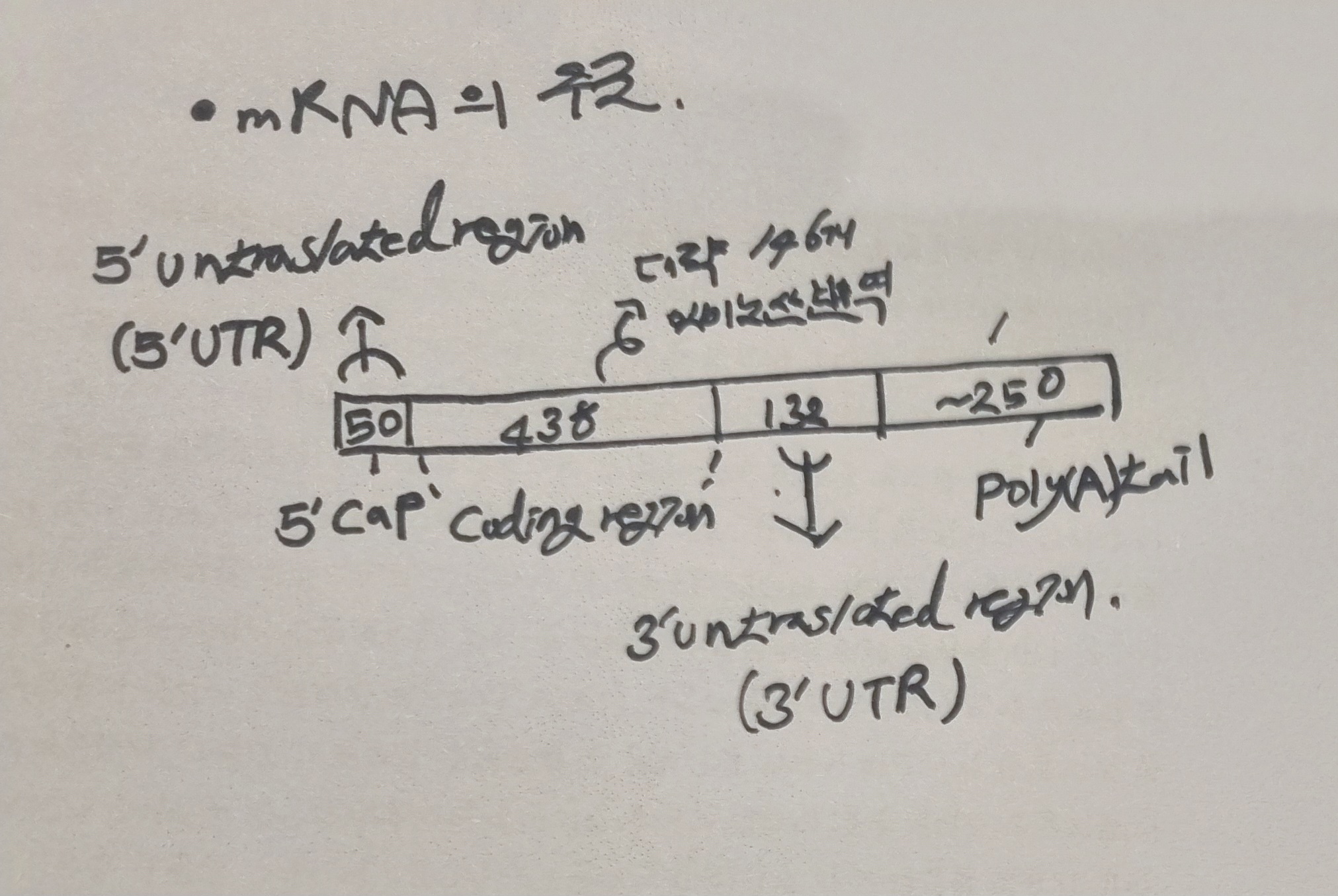
개념이 좀 헷갈리는데 cap & tail 구조가 만들어지기 전의 Primary transcript를 hnRNA(핵에 존재),
cap & tail 구조가 형성되며 intron이 loop 형태가 된게 pre-mRNAs(핵에 존재),
마지막으로 모든 intron이 되고 cap-exon-tail 구조가 되면 mature mRNA(세포질에 존재)로 구분할 수 있습니다.
그렇다면 5' cap과 3' Poly (A) tail이 어떻게 만들어지는지 살펴보겠습니다.
우선, 5' methylguanosine cap이 만들어지는 과정을 영상으로 만들어봤는데, 까페 소음이 같이 들어간 것 같아 양해 부탁드리겠습니다.
글로 설명하자면, 5'의 3개의 phophates(P) 중 마지막 P가 RNA Triphosphatese에 의해 제거되고, 5' terminus는 disphosphates가 됩니다.
이후 guanylyltransferase에 의해 GMP가 inverted orientaion으로 추가되고, 그렇게 RNA의 5'에 있는 P에 5' guanosine이 직면하게 됩니다.
그리고나서 RNA methyltransferase에 의해 7번째 위치의 guanosine base에서 inverted guanosine이 methylated되고, ribose에선 두번째 위치에서 methylated됩니다(그림은 맞지 않을 수 있습니다).
이것이 5' methylguanosine caps process이고 다음으로 3' Poily(A) tails process를 영상으로 만들어보았습니다.
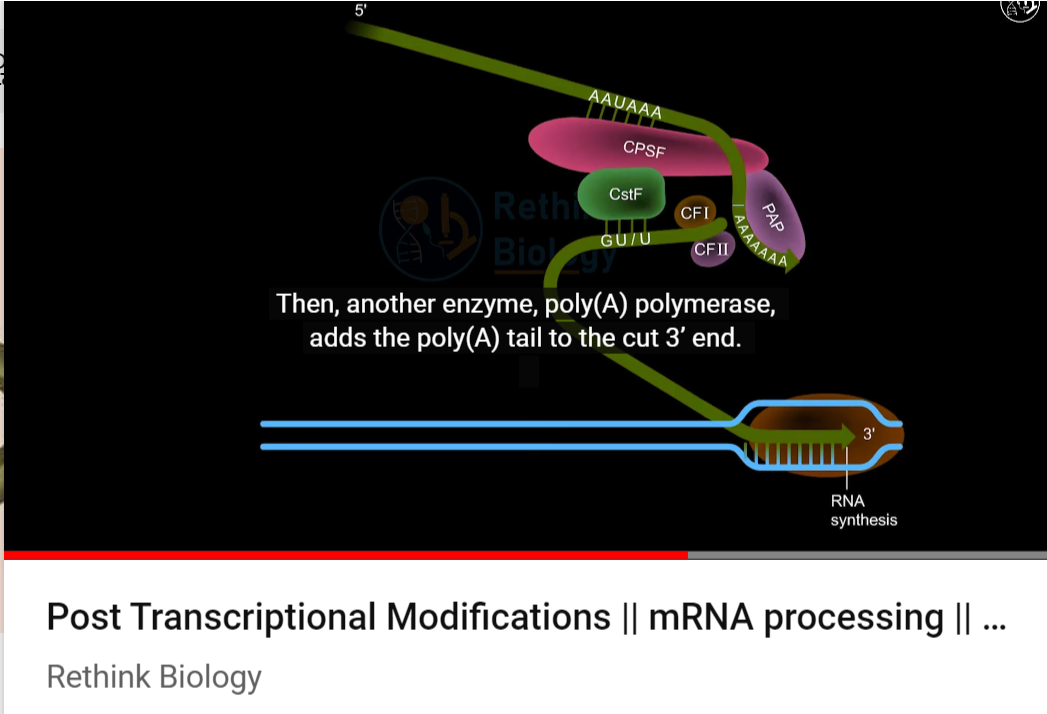
글로 정리하자면, endonuclease와 poly (A) polymerase를 포함하는 Processing complex가 primary transcript를 cleaved하는데,
그러면서 original 3' terminus로부터 새로운 3' end upstream을 생성합니다.
구체적으로 Poly(A) polymerase가 DNA template의 개입 없이 3' end에 AMP의 adenosine residues를 더하고 그러면서 poly (A) tails가 생겨나게 됩니다.
그렇게 5' cap과 poly (A) tail이 생겨난 전사체는 pre-mRNAs가 되게 됩니다. 아래 영상은 꽤 오래된 것이긴 한데 보시면 직관적으로 이해하기 쉽지 않을까 싶습니다.
최근에 나온 아래 영상에서는 5' cap과 3' tailing에 대한 화학적 과정을 소개해주며, 왜 prokaryotic cells는 그러한 절차가 필요 없는지도 알려주고 있습니다.
그 이유는 간단히 말해 원핵생물은 핵이 없으며, 전사와 번역이 동시에 일어나기 때문입니다.
영상을 보니까, 3' tailing을 위한 complex에는 CPSF(개시 site인 AAUAAA 인식), CstF, CFI, CFII와 같은 단백질들이 Poly A polymerase와 함께 3' end를 cut하고 아데노신(A)을 연장하도록 돕는 듯 합니다.
그렇게 만들어진 5' cap은 mRNA의 5' end가 exonucleases에 의해 소화되는 것을 막을 뿐만 아니라, 리보솜이 세포질에 있는 mRNA를 인식하고 결합하도록 돕습니다.
그리고 poly A tail은 mRNA를 안정화하여 세포질에 있는 효소들로부터 degraded되는 것을 막습니다.
참고로, 포유류의 mRNA는 200 ~ 250개 정도의 adenosine residues를 포함한다고 합니다.
다음 글에서는 pre-mrRNAs에서 Introns를 제거하는 RNA splicing에 대해 알아보도록 하겠습니다.




