이번 글에서는 전 글에 이어 전공 수업 때 배운 'Transcription' 관련 이론 복습을 해보려고 하는데
먼저 bacteria의 Transcription 과정을 정리해보겠습니다.

[대학원 준비 4일차] 이론 복습 :: RNA polymerase에 의한 transcription process
들어가기 전에 우선 Eric Chow라는 분이 NGS methods에 대해 잘 설명해주시는 것 같아서 아래 설명 영상도 어제 저녁에 봤는데, NGS(next generation sequencing)란 무엇이고 어디에 적용될 수 있는지 overview가
tkmstudy.tistory.com
RNA polymerase는 우선 5개의 subunits이 core enzyme을 형성하는데,
이 효소는 accessory polupeptide인 signma factor (σ)가 promoter site의 효소 affinity를 높인 덕분에 분리된 DNA 가닥에 붙게 됩니다.
이러한 분리된 DNA, sigma factor, polymerase의 복합체를 'Open Complex'라고 합니다.
10개 정도의 nucleotides가 incorporated된 후 효소는 conformational change를 겪어 transcriptional elongation complex가 되어 움직입니다.
참고로 RNA polymerase에는 Primer를 필요로 하지 않는다는 특징을 갖습니다.
위의 영상을 보시면 아시겠지만 RNA 분자의 5' end를 향하는 부분을 upstream,
3' end를 향하는 부분을 downstream이라고 합니다.
그리고 첫번째로 생겨난 염기쌍에 '+1'에 위치하게 됩니다.
위치를 기반으로 설명하자면 Sigma factor는 nontemplate strand에 개시부위 10 앞쪽(-10)의 conserved sequecne인 TATAAT로된 Pribnow box를 익식하고 상호작용하여 promoter DNA의 인근 부위를 melting합니다.
그 덕분에 RNA polymerase가 primer의 필요 없이 promoter에 붙을 수 있는 것입니다.
해당 지역(TATAAT)은 두개의 DNA 가닥 사이에 hydrogen bonding이 매우 약해서 RNA polymerase에 의해 쉽게 분리된다1)합니다.
아래 영상에선 RNA polymerase의 subunits에 대한 설명이나오고, 뒷부분에 Termination(Rho dependant와 indenpendant)에 대한 얘기가 나옵니다.
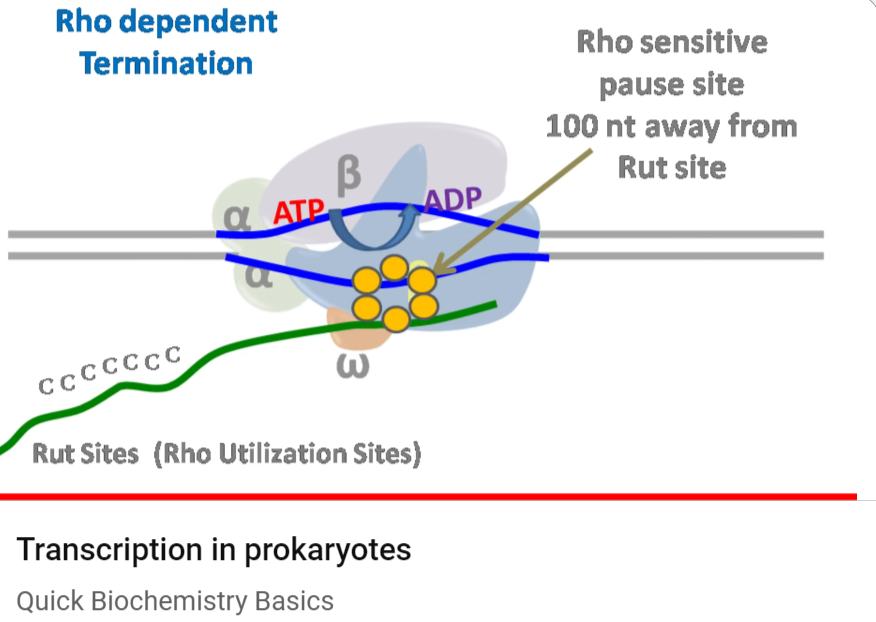
정리하자면, 전사의 Termination은 'Rho'라는 단백질에 의존하지 않느냐 의존하느냐에 따라 구분되는데,
Rho independant termination의 경우 GC-rich rigeon이 hairpin 구조를 형성하여 RNA polymerase를 멈추게 한 후
약한 RNA-DNA hybrid를 형성하는 U-rich sequence가 RNA transcript를 DNA로부터 떨어지게 만들어 전사는 종결됩니다.

Rho에 의존하는 termination의 경우 Rho가 새로 합성된 RNA를 둘러싸며 5' -> 3'으로 움직여 polymerase로 가서
RNA transcript를 DNA로 부터 떨어뜨려 종결이 이루어지게 됩니다.
그럼 이제 prokaryotic cells(원핵세포)를넘어 Eukaryotic cells에 대해 다뤄보려고 합니다.
그전에 앞서 등장했던 RNA polymerase에 대해 더 자세히 알아보도록 하겠습니다.
우선 Eukaryotes에는 3가지 타입의 RNA polymerases가 있는데요, 대부분의 rRNAs를 전사하는 RNA polymerase I,
mRNAs를 전사하는 RNA polymerase II, tRNAs를 전사하는 RNA polymerase III가 있습니다.
참고로 microRNA와 snRNA는 RNA polymerase II가 만듭니다.
이렇게 전사된 세 가지 타입의 eukaryotic RNAs는 처음엔 final RNA product보다 상당히 깁니다.
이러한 initial RNA product를 'pimary transcript(or pre-RNA)'라고 하고 "cut-and-paste"를 거쳐 더 작은 functional RNAs(90 ~ 100 nt) 사이즈가 됩니다.
우선 리보솜을 만드는 rRNA의 synthesis와 processing 절차에 대해 정리해보겠습니다.
eukaryotic cell은 엄청난 양의 ribosomes를 포함한 만큼 수많은 양의 rRNA(ribosomal RNA)를 포함합니다.
따라서 대부분의 세포에서 80% 이상의 RNA는 rRNA입니다.
rRNA를 인코딩하는 DNA sequences는 rDNA라고 하는데, 이는 수백 번정도 repeated되고, ribosome production의 효율을 높일 수 있도록 genome의 하나 혹은 일부 지역에서 clustered된다고 합니다.
또한 분화되지 않는 세포에서, 이 rDNA는 서로 모여 하나 혹은 그 이상의 'nucleoli(singular nucleous)'라 불리는 비정상적인 모양의 nuclear structure를 만들고, 결국 이것이 ribosomes를 만들게 됩니다.
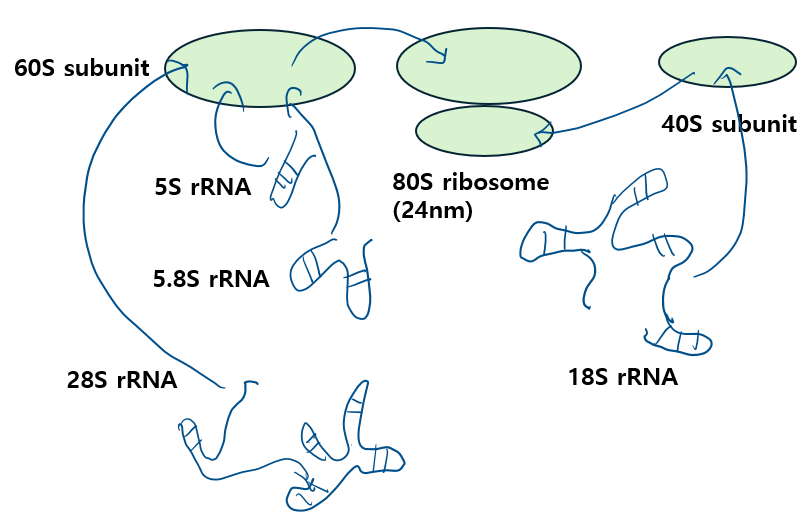
Eukaryotic ribosomes는 4개의 구별되는 ribosomal RNAunits를 가지는데
3개는 28S, 18S, 5.8S라는 large subunit이고, 다른 하나는 5S의 조그만 subunit입니다.
여기서 세가지 rRNAs(28S, 18S, 5.8S)는 RNA polymerase I에 의해 합성된 pre-rRNA로부터 다양한 nucleases에 의해 carved되고,
5S rRNA는 RNA polymerase III에 의해 seperate RNA precursor로부터 합성된다고 합니다.
중요한건 pre-RNA가 먼저 cleaved되기 전까지, methyl groups는 ribos groups에 더해지고,
95%의 uridine residues pseudouridine으로 전환됩니다. 이를 'post transcriptional modification'이라고 합니다. 매우 중요한 개념이지만 다음에 더 자세히 다루도록 하겠습니다.
먼저 어떻게 18S, 28S, 5.8S, 28S, 그리고 5S가 만들어지는지는 아래 영상을 참고하시면 될 것 같습니다.
참고로, pre-RNA processing은 small, nucleolar RNAs(snoRNAs)의 도움에 의해 이루어집니다.
그리고 snoRNAs는 packed된 단백질들과 함께 small, nucleolar ribonucleoproteiins(snoRNPs)를 이룹니다.
snoRNPs는 rRNA의 pseudouridylation*나 2′-O-methylation*와 같은 ribosomes biogenesis 역할을 하며,
pre-rRNA processing을 가이드하고, molecular charpeones로서 기능을 하게 합니다3).
pseudouridylation : uridine을 pseudouridine으로 전환해 RNA 구조 안정화
2′-O-methylation : ribose suger에 methyl group을 더하여 유전자 발현 조절
구체적으로 box C/D snoRNAs (U20 snoRNA)는 pre-rRNA에 있는 nucleotides 중 어떤 ribose groups를 메틸화할지 결정하고,
box H/ACA snoRNAs(U68 snoRNAs)는 어떤 uridines를 pesudouridines로 전환할지 결정합니다.
그리고 이 두가지의 snoRNAs 모두 rRNA에 상보적인 10~21 nt 길이의 긴 RNA-RNA duplexs를 만듭니다.
그렇게 snoRNA가 효소를 가이드해서 methlyase와 pseuoduridylase가 이루어지도록 특정 뉴클레오티드를 변형시키는 것인데 그것에 대한 자세한 정리는 시간 관계상 다음 글에서 해보도록 하겠습니다.
참고자료
1) Oxford Reference, Pribnow box, A dictionary of Biology, URL : https://en.dict.naver.com/#/search?range=all&query=prokaryotic
2) John W. Pelley PhD, 16 -RNA Transcription and Control of Gene Expression, Elsevier's Integrated Biochemistry, p 135~145 (2007).
3) Ojha S, Malla S, Lyons SM. snoRNPs: Functions in Ribosome Biogenesis. Biomolecules. 10(5):783. ( 2020).

'생물정보학(바이오인포매틱스)' 카테고리의 다른 글
| [대학원 준비 5일차] transcription in eukaryotes, 5' methyIguanosine cap(5' capping)과 3 (1) | 2024.09.24 |
|---|---|
| [대학원 준비 5일차] 이론 복습 :: U20 snoRNA, U68 snoRNA, 그리고 tRNA(transfer RNA) (0) | 2024.09.24 |
| [대학원 준비 4일차] 이론 복습 :: RNA polymerase에 의한 transcription process (4) | 2024.09.22 |
| [대학원 준비 3일차] SMART-seq에 대하여 (template switch) (4) | 2024.09.21 |
| [대학원 준비 3일차] single-cell RNA sequencing (scRNA-seq)에 대하여 (0) | 2024.09.21 |



