이번 글에선 최근에 개별세포분석 리뷰논문을 읽으면서 관련 툴들을 노션에 정리하다 찾게 된 흥미로운 툴인 CellTypist에 대해 소개해보겠습니다.
대학원 준비생의 노트 ( 24. 12 ~ ) | Notion
'과학커뮤니케이터 TKM' TKM's STUDY BLOG
sparkling-dibble-7ff.notion.site
CellTypist는 자동화된 cell type annotation을 위한 open source tool로 다음과 같은 working group에 해당 모델을 개발했다고 합니다.

한국인 분도 계신 듯 하여 반갑네요. 아래는 CellTypist 플랫폼 링크입니다.
Automated cell type annotation for scRNA-seq datasets
CellTypist provides automated cell type annotation for scRNA-seq datasets
www.celltypist.org
위의 링크로 들어가면 다음과 같은 화면이 등장할겁니다.

왼쪽 버튼들을 보니까 dataset를 불러오면 바로 cell-type을 annotation해주는 것 같고, tutorials도 제공해주는 듯 하며, cell-type encyclopedia라는 것도 있네요. 하나씩 알아보겠습니다.
1. Annotate your dataset now
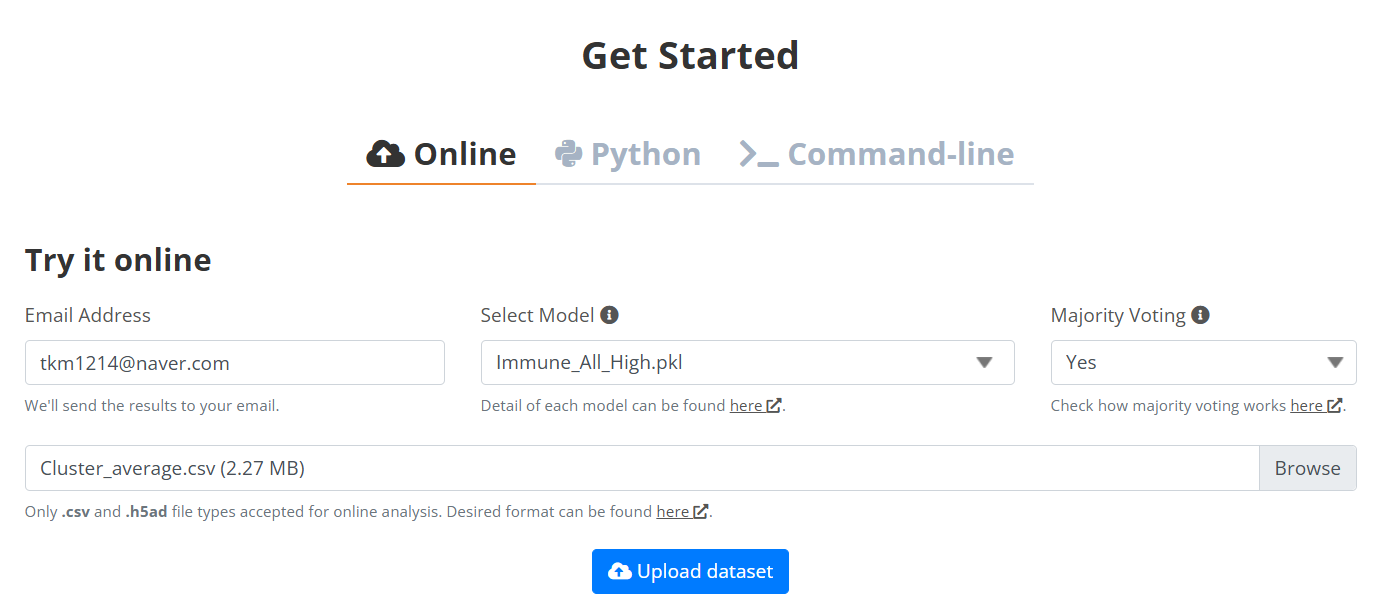
해당 버튼을 누르면, 요런 화면이 등장할텐데요. 결과를 받을 이메일 주소를 입력하고, cell-type annotation을 진행할 파일(csv or h5ad 형식)을 지정한 후 참조할 Model을 선택하고, Majority voting을 할지 말지 결정하면 되는 듯 합니다.

Get started 아래 Online 말고도 'Python' 혹은 '>_Command-line'을 선택해서 관련 코드를 복사한 뒤 파이썬 혹은 conda 환경에서 celltypist 패키지를 import해서 활용할 수도 있는 듯 합니다.

저는 Online 메뉴에서 저번에 Seurat로 scRNA-seq 데이터를 분석할 때 받은 csv 파일을 넣어 annotation을 진행해보도록 하겠습니다. 궁금하신 분은 아래 링크를 확인하시면 되겠습니다.
[25일차] BRIC의 scRNA-seq data 분석법 글 따라해보기 04 :: VlnPlot, BoxPlot, DotPlot, Cluster Annotiation
전 글에 이어서 이번에는 필터링하고 PCA 및 클러스터링한 PBMC 데이터를 가지고 또 다른 Plot들을 그려보도록 하겠습니다. 동향 | BRIC" data-og-description="1. 들어가며 R을 활" data-og-host="tkmstudy.tistory
tkmstudy.tistory.com
분석을 진행할 파일은 요렇게 각 cluster에서 HVGs transcript의 발현량이 얼마인지 나타나 있는 csv 파일(Cluster_average.csv)입니다.

csv 파일을 넣고 이메일 주소를 기입하고, model을 선택하고자 하는데요. model 선택을 위해 model에 대해 설명해주는 아래 'here'를 클릭했습니다.

그럼 모델 선택을 위해선 query data의 context를 기준으로 선택하라는 말과 함께 model list에 대한 자세한 설명이 나오는데요, 제가 PBMC(10k Peripheral Blood Mononuclear Cells) 데이터를 사용했던 만큼 아래 적힌 Adult_COVID19_PBMC를 모델로 사용할까 했지만 설명을 읽어보니 먼저 Immune_All_Low/High 모델을 사용하길 추천해서 해당 모델을 먼저 돌려보고, Adult_COVID19_PBMC도 돌려보겠습니다.
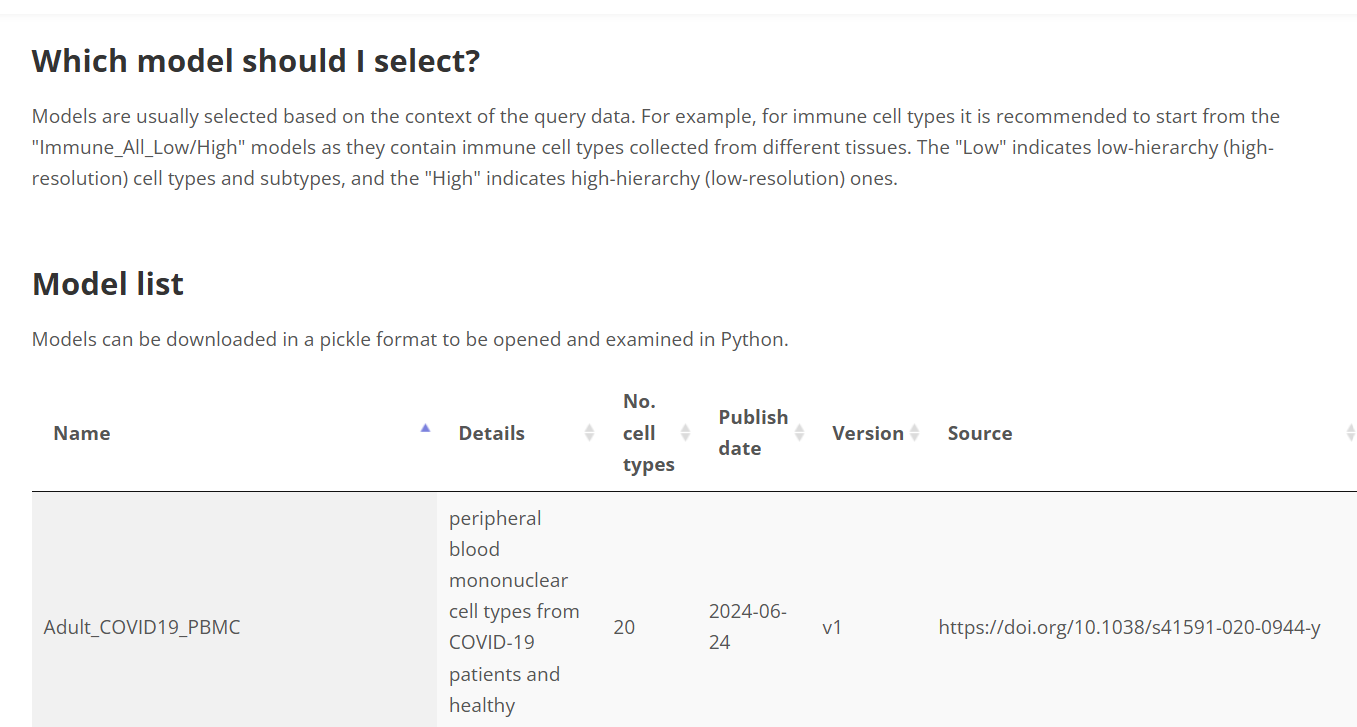
Immune_All_Low/High 중 Low는 low-hierarchy로 high resolution, High는 high-hierarchy로 low resolution이라고 하는데요. 챗GPT한테 물어보니 특정 세포 유형의 희귀한 하위집단(ex. 특정 T 세포 아형(CD4+ Tfh, Th1, Th2)을 구분)을 찾고자 할땐 Low, 일반적인 면역 반응의 경향성을 파악하거나 초기 단계의 데이터 탐색(T 세포, B 세포, 단핵구와 같은 주요 면역 계열로 데이터를 요약)할 땐 High가 유용하다 길래, High로 선택해보겠습니다.

그럼 이제 Try 하기전 마지막 선택 요소인 major voting을 할지 말지 결정하고자 하는데요. major voting은 prediction result를 정제해주지만 runtime을 증가시킨다고 합니다. 그냥 yes로 하고 너무 오래걸린다 싶으면 취소하고 다시 no로 해보겠습니다. 이제 'Upload dataset'을 클릭해보겠습니다.
좀 기다리다보면 요렇게 all good! 이라면서 입력한 메일 주소로 파일을 보냈다고 말해줍니다.

이메일을 확인하니 요런 메일이 와있었습니다. 주소를 클릭해보겠습니다.

그러면 요런 파일이 다운받아지는데요.
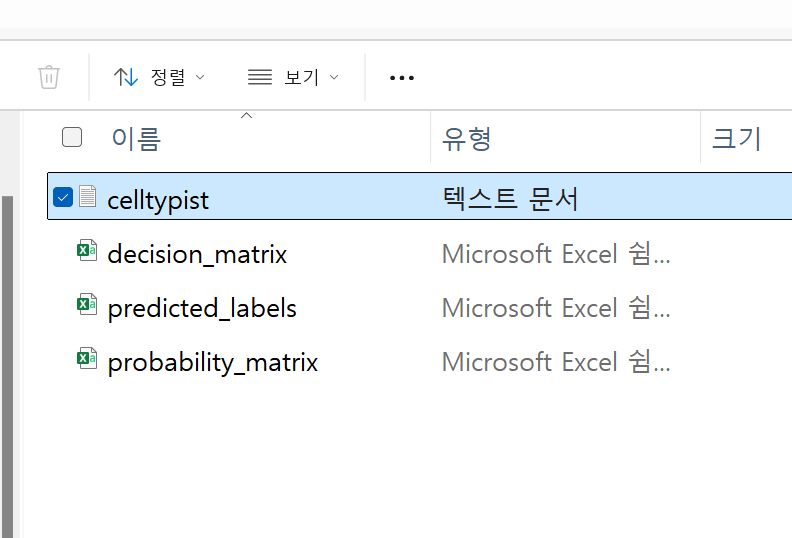
그중에서 요렇게 predicted labels를 열면 각 클러스터가 어떤 세포인지 annotation한 결과값이 나옵니다.

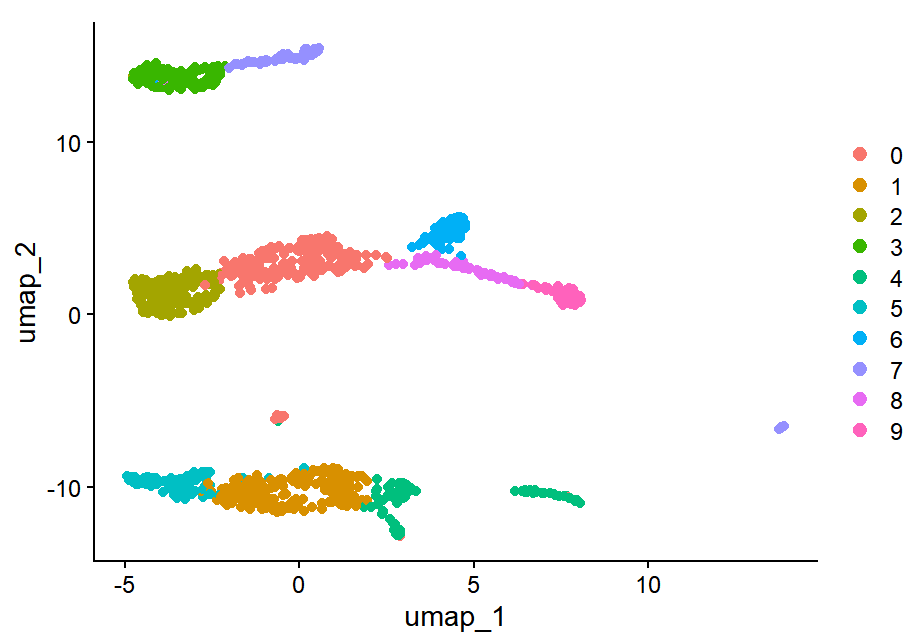
참고로 9개의 클러스터를 UMAP에 시각화한 plot은 다음과 같습니다.
다음으로 Adult_COVID19_PBMC 모델로도 동일판 파일로 돌려봤는데, 오류가 떴습니다.
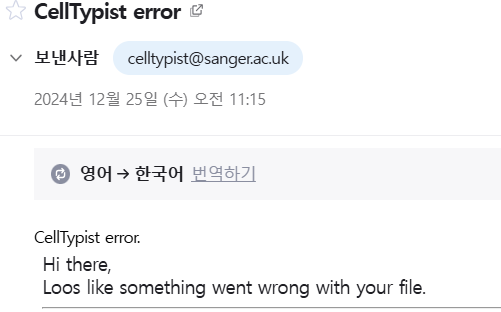
이게 COVID-19라는 조건이 붙어 있는 만큼 제가 사용한 기본적인 PBMC를 cluster annotation하기엔 적합하지 않았나 봅니다. Immune_All_High로 label을 확인했으니 넘어가도록 하겠습니다.
이번엔 High resolution인 Immune_All_low로 돌려봤는데 다음과 같은 label이 나왔습니다. resolution이 높은 만큼 보다 구체적으로 알려주는 듯 합니다.

1에서 8까진 low와 high가 상위와 하위 분류 이런 식으로 같은 세포로 나타났는데, 클러스터 9의 경우엔 low에는 ILC(선천성 면역세포), high의 경우엔 CD16+ NK cells로 뭔가 달라보여서 이게 뭔지 확인해봤는데요. 챗GPT한테 물어보니까 CD16+ NK cell이 ILC1의 하위분류라고 하네요.

2. Tutorials
첫 화면 Tutorial 버튼을 클릭하면, 요렇게 Online User's Guide와 진행할 수 있는 세 개의 tutorial이 나오는데요. 그중 interative tutorial (I)을 클릭해보겠습니다.
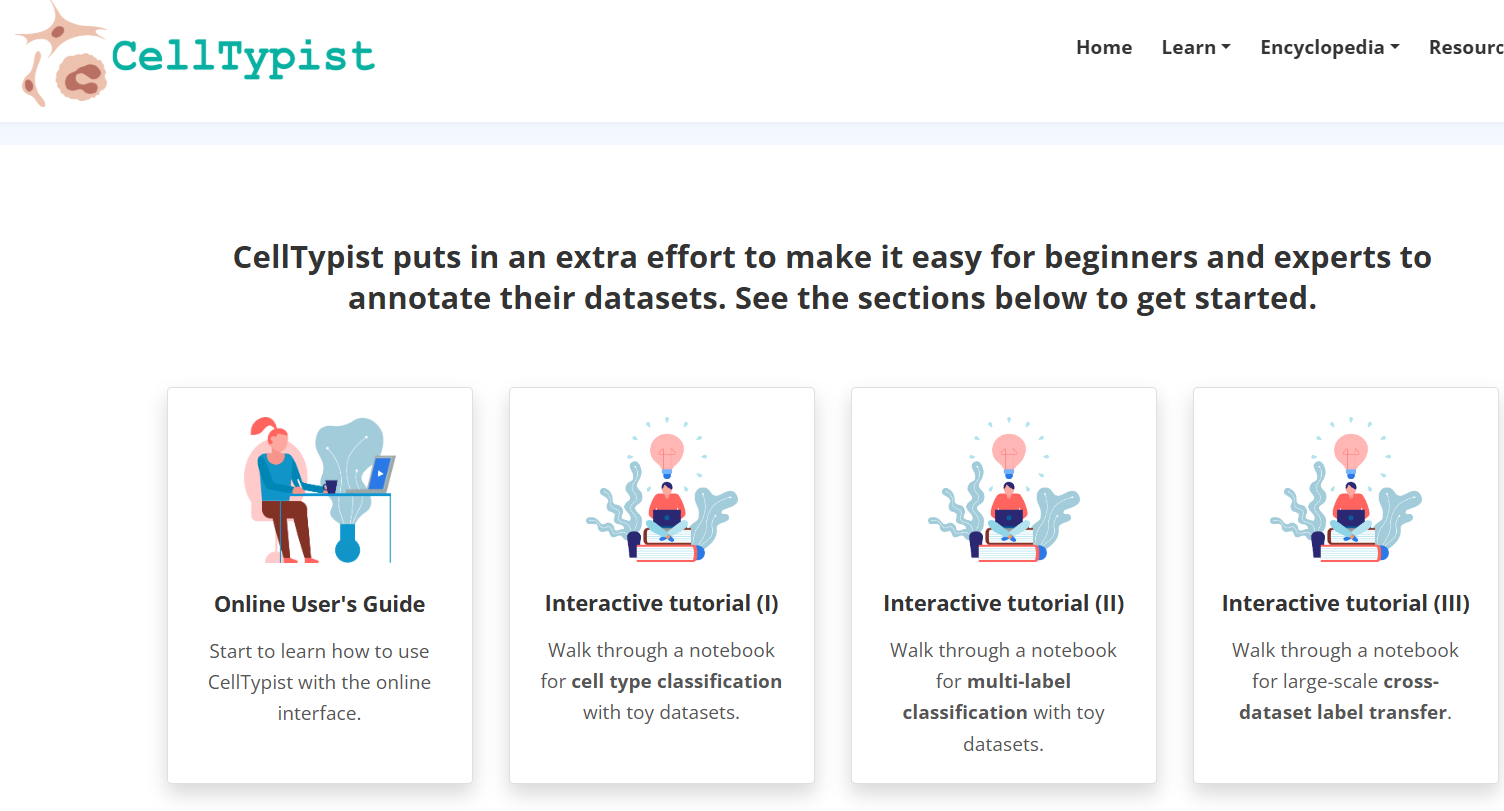
클릭을 하게 되면 아래와 같이 구글 코랩 노트북이 등장하면서 scanpy 패키지와 celltypist 패키지를 임포트해서 cell-type classification을 진행할 수 있는 코드가 나옵니다.

참고로 최근에 Scanpy tutorial 코드 한 줄 한 줄 해석보는 글을 티스토리 블로그에 다가 비공개로 쓰고 있는데요.
현재 clustering까지 학습한지라 다음 cell-type annotation 단계에서 요 cellTypist를 쓰지 않을까 싶습니다. 안쓰면 따로 해보겠습니다.
Preprocessing and clustering
The data used in this basic preprocessing and clustering tutorial was collected from bone marrow mononuclear cells of healthy human donors and was part of openproblem’s NeurIPS 2021 benchmarking da...
scanpy.readthedocs.io
각 Cell type의 canonical marker genes, Genes와 관련된 Cell-type을 알려주는 사전인 듯 합니다. 아래는 Immun v2인데, 오른쪽 상단에 Encylclopedia를 눌러 Immune cells 말고 Embryonic cells를 확인할 수도 있습니다.

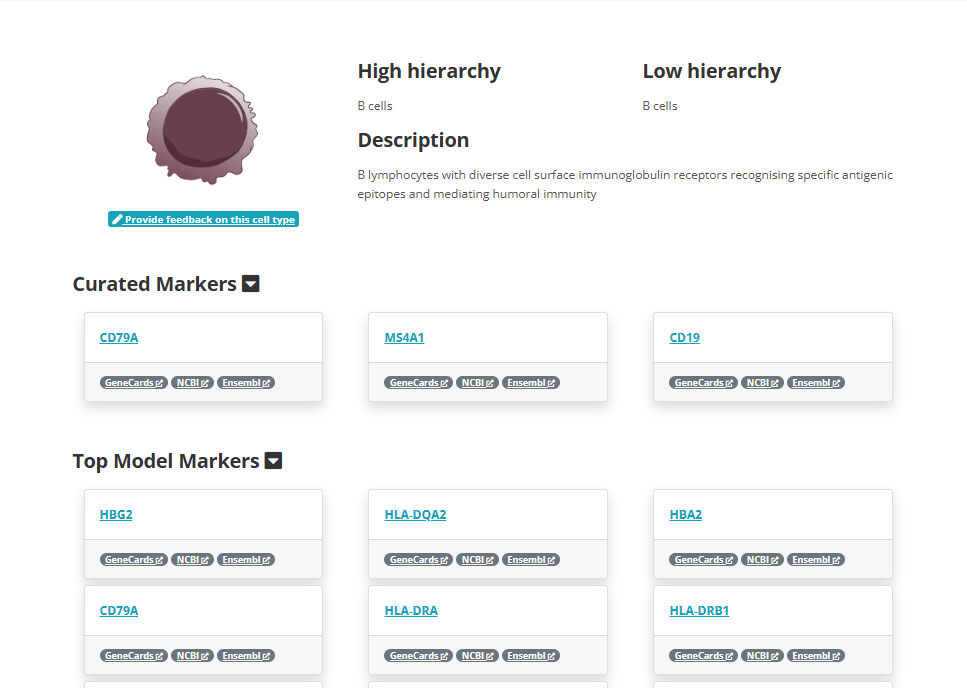
저는 여기서 먼저 Cell types의 B cells를 눌러봤습니다. 그럼 이렇게 짧은 설명과 함께 curated markers와 Top Model Markers가 무엇인지 나타내줍니다.
Cell-type 말고 이렇게 유전자를 지정해서 해당 유전자가 어떤 기관(ex. 간, 눈 등), 어떤 세포(ex T cell, B cell 등)에서 주로 등장하는지 히트맵으로 확인할 수도 있습니다.

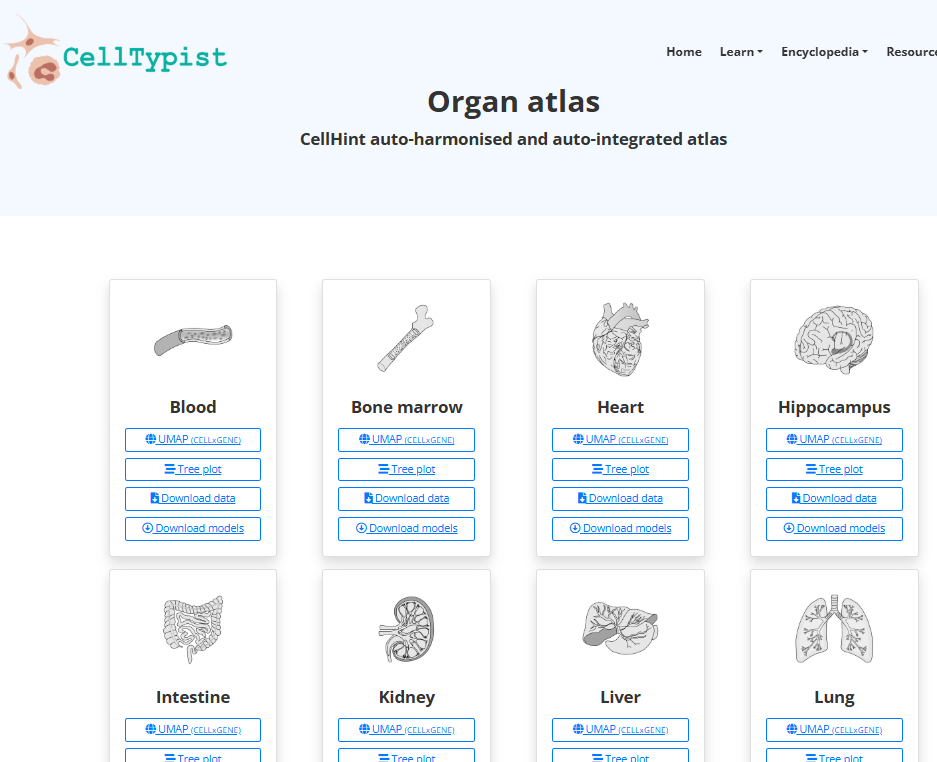
이외에도 Celltypist에는 Organ atalas도 요렇게 정리해주는데요, 글이 길어지니 궁금하신 분들은 해당 링크를 클릭해서 확인해보시길 추천드립니다. 감사합니다!
참고자료
1) CellTypist, URL : https://www.celltypist.org/
'생물정보학(바이오인포매틱스)' 카테고리의 다른 글
| [31일차] 생물학 전공자를 위한 오래된 유튜브 추천, z집사TV(ZipsaTV) (0) | 2025.01.28 |
|---|---|
| [30일차] 논문 간단 소개 :: 장내 미생물이 면역계, 그리고 궤양성 대장염(ulcerative colitis)에 미치는 영향 (3) | 2024.12.29 |
| [28일차] Azimuth, reference-base single-cell analysis를 위한 앱 (3) | 2024.12.22 |
| [27일차] Git & Github 기초 공부 06 :: 깃허브 원격 저장소와 로컬 저장소 연결, remote + push & pull (2) | 2024.12.21 |
| [27일차] Git & Github 기초 공부 05 :: 브랜치 관리하기 :: 실험용 브랜치 생성, 병합, 제거까지 (2) | 2024.12.21 |



