전 글에서 말씀드렸듯이, 요즘 Zotero를 활용해 논문 하나를 제대로 분석해보는 연습을 하고 있습니다.
[11일차] 대학원 준비생을 위한 'Zotero'를 활용한 논문 학습 방법
안녕하세요, 오늘은 어제부터 활용하고 있는 'Zotero'라는 논문 정리 플랫폼 활용 방법을 정리해보려고 합니다. Zotero | Your personal research assistant www.zotero.org 사용한지 하루 밖에 되지 않아 활
tkmstudy.tistory.com
연구가 익숙치 않다보니 한 줄 한 줄 이게 뭔지, 그리고 왜 활용되고 어떻게 적용되는지 알아가고 있는지라 논문 하나 읽는데 꽤 시간이 걸릴 느낌입니다.
물론 논문 하나만 본다기보다 논문에 나온 개념과 관련한 논문, 자료 조사, 선행 논문 등을 찾아보면서 공부하고 있습니다.
그렇게 하다 보면 논문 하나 읽어도 알아가는게 많지 않을까 싶습니다. 그런 차원에서 논문에 나온 개념 몇가지만 오늘 글에서 정리하고 가도록 하겠습니다.
1. TetO(Tet operator)
Operator라고 하니 전에 bacteria에서의 Lac operon의 개념에 대해 정리해봤던 기억이 나는데요, 그때 regulatory elements로 operator, promoter 등이 있었죠. Tet operon도 operon이니 개념에 있어 유사한 부분이 있습니다.
[10일차] Lac Operon(structural genes, promoter, operator), 그리고 lactose와 관련된 β-galactosidase 등의 효소
오늘은 Translation 관련 이론 복습에 이어 Gene Regulation 관련 이론 복습을 해보도록 하겠습니다. [9일차] Translation 과정 :: Initiation, Elongation, Termination 그리고 Nonsense Mutation오늘 글부터는 Translation
tkmstudy.tistory.com
Tet operon은 tetracyline*-derivatives인 doxycyline(dox)*가 투입되면, 이것이 Tet repressor(TetR)에 붙어 conformational change를 야기하고, repressor가 tetO(tet operator)에 결합하지 못하게 되어 유전자 발현이 활성화1)된다고 합니다.
* 테트라사이클린(tetracyline) : 세균에 의한 감염을 치료하는 항생제로, 세균의 단백질 합성을 저해함0).
* 독시사이클린(doxycycline) : 세균에 의한 감염을 치료하는 항생제로 테트라사이클린의 구조를 일부 변형하여 개발. 테트라사이클린보다 작용 시간이 길어 투여 횟수가 적은 장점이 있음0).
참고로 Bacterial Tet Operon의 regulartory region은 promoters PA와 PR1/PR2가 overapping 되어있고, 그곳에 두 개의 tetO operator sequences가 존재합니다.
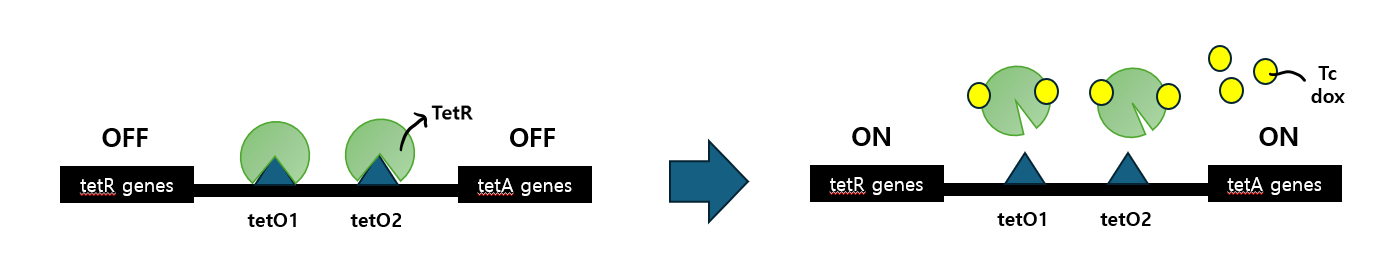
이러한 operon의 특성을 이용하여 eukaryotic cell에서 유전자 활성을 조절할 수 있는 tetracycline-controlled Tet-Off와 Tet-On gene expression system이라는 생명공학 기술이 개발되게 되는데요, 하나씩 소개해보겠습니다.
1-1. Eukaryotic Tet-off system
선행 논문1)에 따르면, Tet-Off system에서는 virus VP16 protein의 transcription activation domain (AD)에 207-aa TetR(Tet repressor)이 융합된 tetracycline-controlled transcriptional activator(tTA)가 활용된다고 합니다.
이게 TeR이 있다고 해서 Operator에 결합하면 유전자 발현을 억제한다고 생각하기 쉽지만, TetR이 있어 본 융합체가 operator에 결합하면서도 VP16이라는 전사 활성 영역이 융합체가 transcription activator 성격을 나타내게 한다는 걸 기억해야 합니다.
이때 Eukaryotic Tet-off system인 만큼 TATA-box와 tetO sequences로 구성된 Tet promoter(Ptet)에 tTA가 결합하게 되면 gene of interest(G.O.I)의 expression을 활성화하게 됩니다.
그러나 Tc나 dox를 주입하여 이것이 tTA에 결합하게 되면, 아래 이미지처럼 tTA의 TetR domain에 conformational change가 발생하고, tRA가 tetO binding을 하지 못해 유전자 발현이 비활성화됩니다.
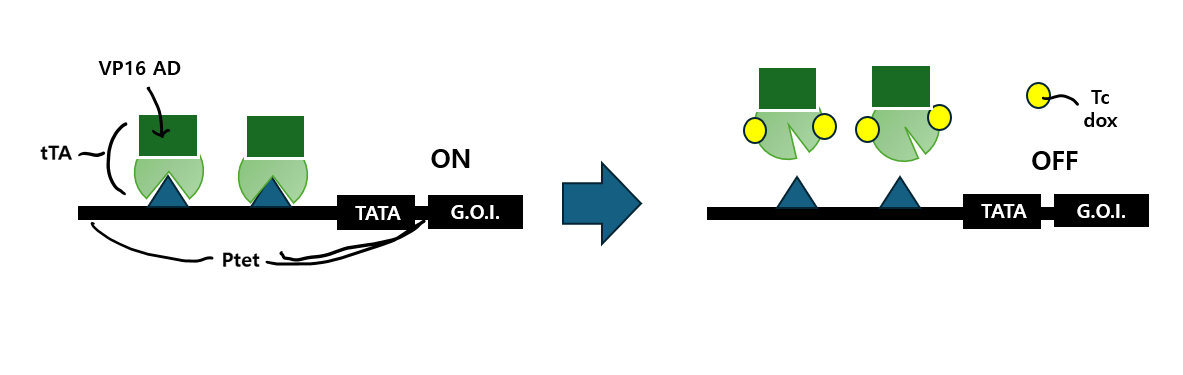
이렇게 Tc나 Dox의 존재에 따라 promoter에 tTA transcription activator의 결합이 억제되는 생물학적 시스템2)을 Tet-Off system이라고 합니다.
그러나 선행 논문1)에 따르면, Tet-off system은 Tc 혹은 Dox를 제한된 시간 내에 지속적으로 넣어줘야 하고, 즉 유전자 발현의 활성이 불 필요할 때 지속적으로 effector를 넣어주어야 하고, 그렇게 진행되는 지속적인 노출이 실험 결과를 도출함에 있어 바람직하지 못할 수 있다고 합니다.
무엇보다 gene expression 활성을 하기 위해 tetracyline을 제거하기 위한 절차가 필요하는데, 물론 세포를 washing하거나 culture medium의 교체를 통해 간단히 제거할 수 있긴 하지만, large cell cultures를 사용할 땐 제거하는게 쉽지 않다 합니다. 그래서 등장한게 Tet-on system입니다.
1-2. Eukaryotic Tet-on system
Tet-on-system은 dox를 더함으로써 비활성화되어있던 gene expression을 활성화할 수 있는 시스템입니다.
여기에는 effector가 존재할 때 tetO에 결합하는 TetR variant인 reverse-TetR(rTetR)이 역할을 하며, 이는 original protein(TetR)과 4개의 아미노산 position이 다르다1)고 합니다.
즉, dox의 rtTA 결합이 rtTA의 conformational switch를 유발하고 TetO binding을 허락하여 GOI에서 유전자 발현이 활성화되게 됩니다.
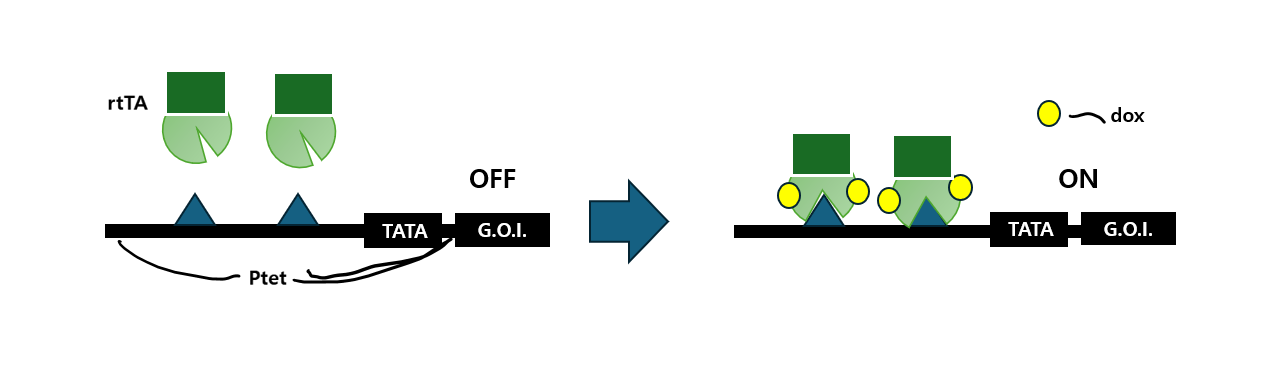
두 가지 Tet system에 대한 소개와 이를 활용해 target gene을 control하는데 활용하는 방법(간단히 정리하자면 TRE(Tetracycline response element) vector를 먼저 주입하고, tTA 혹은 rtTA를 포함한 Vector로 실험 control)에 대한 소개는 아래 영상에 잘 설명해주고 있으니 참고바랍니다.
참고로 현재 공부하고 있는 논문에선 reprogamming genes(G.O.I)를 위한 cassette가 될 수 있는 ColA1(TetO-OSKM) ; ROSA26(rtTA) mice를 활용했습니다(Tet-on system).
2. 2A PEPTIDE
2A peptide는 single mRNA로부터 multiple genes를 co-expression하기 위해 사용하는 방법인데요, 관찰하고자 하는 유전자가 세포에서 잘 발현되었는지 검출하기 위해, G.O.I(gene of interest)와 함께 reporter(fluorescence gene or resistance gene)을 co-expression할 수 있는 것은 물론, 같은 cassettes에서 plasmide에 있는 multiple genes를 발현하게 하는데에도 유용하다3)고 합니다.
사실 single transcript로부터 multiple genes를 번역하기 위해 사용하기 위해 먼저 나온 방법은 virus의 mechinary인 IRES(Internal Ribosome Entry Site) element를 eukaryotes에 활용하는 방법으로, IRES는 말 그대로 단백질을 합성하기 위해 ribosomes를 recruit하는 기능을 가진 RNA sequences4)입니다.
보통 진핵세포에서 리보솜은 mRNAs의 5' cap에서 전사를 시작하는데(cap-dependent mechanism), strong cellular stresses가 가해지는 특정 조건(ex. viral infection)에선 virus의 IRES가 host cell의 translational machinery이자 cap-independant mechanism로서 붙어서 붙은 특정 region에 리보솜을 불러와서 번역을 개시2)해 아래 이미지처럼 single mRNA로부터 두개의 단백질을 coexpression할 수 있습니다.
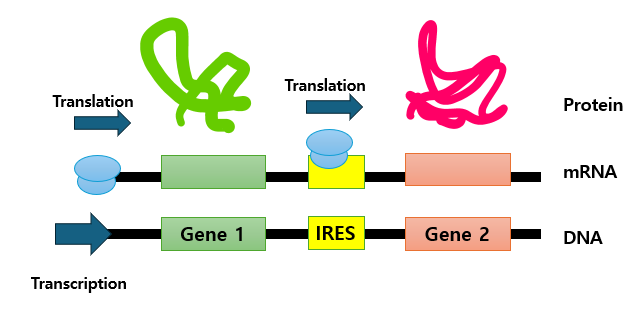
아래 영상에서는 Eukaryotes와 Bacteria에서 다르게 일어나는 번역 과정, IRES에 의한 예외적인 번역 과정, 이 IRES를 활용해 2개의 genes를 하나의 eukaryotic mRNA에서 발현하는 과정에 대해 하나하나 잘 설명해주고 있으니 참고 바랍니다.
그렇지만, IRES elements는 꽤 커서(500~600 bp) vrial transfer vector에 담기 어려울 수 있고, 두 개보다 많은 genes를 발현할 수 없다는 한계3)가 있다고 합니다. 이러한 IRES elements의 한계를 극복하기 위해 등장한 것이 바로 '2A peptides'입니다.
2A peptides는 eukaryotic cell의 번역 과정에서 polypetides의 'cleavage'를 매개하는 18~22 amino-acid-long viral oligopeptides로,
2A의 C-terminus에서 glcyl-proyl peptide bond를 형성하는 과정을 리보솜이 자연적으로 skip하는 2A-meidated self-cleavage mechanism을 인위적으로 활용하여 하나의 mRNA로부터 multiple gene을 co-expression하는 기술5)로 적용될 수 있으며, 많은 transgenes를 발현시키는데 IRES보다 유용하다고 합니다.
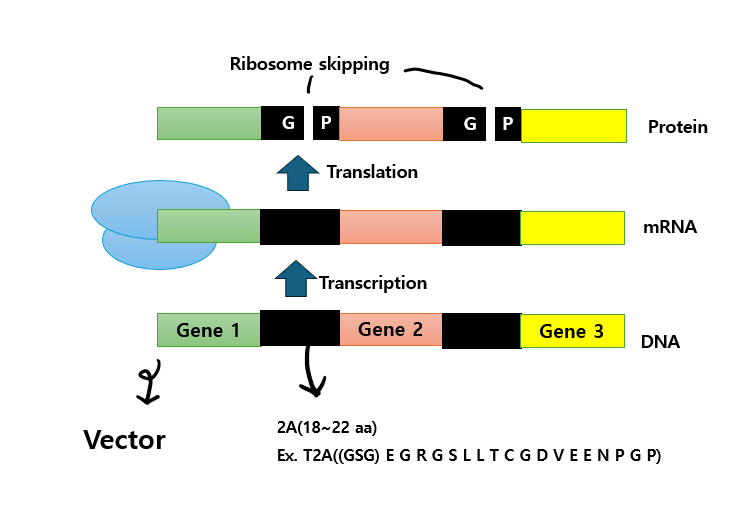
참고로, 2A peptides는 G.O.I 사이에서 PCR-cloned*될 수 있고, 그럼으로서 single unite으로서 backbone에 전체의 casatte를 삽입할 수 있다3)는 장점이 있습니다.
* PCR cloning: PCR product를 붙잡아서 vector of interest로 만드는 것6).
IRES와 2A의 장단점을 정리한 Vector Builder 글7)에 따르면, IRES는 2A와 달리 upstream 또는 downstream ORF(open reading frame, 단백질이 합성될 잠재력이 있는 region)의 단백질의 서열에 영향을 주지 않지만, downstream ORF가 upstream ORF에 비해 아주 낮은 수준으로 발현되며, 앞서 말했듯 사이즈가 커서 패키징이 쉽지 않고 transgenes의 수가 제한된다는 단점이 있다고 합니다.
2A는 ORF의 단백질의 서열에 영향을 주어 원치 않는 생물학적 효과를 발생시킬 수 있고, 2A self-cleavage가 100% 효율적이진 않다는 단점이 있지만, downstream ORF가 upstream ORF에 비해 상당(혹은 약간 낮은) 수준으로 발현될 수 있다는 장점이 있다고 합니다. 결국, 두번째 ORF(ex. drug-selection marker)를 높은 수준으로 발현할 필요가 없으면 IRES, ORF가 두개 이상이거나 ORF 발현이 동등 수준이어야 할 땐 2A peptide가 유용하다고 합니다.
Vector Builder 플랫폼을 보니까 제가 직접 Vector를 설계해보는 시뮬레이션을 해볼 수 있는 것 같은데, 나중에 한번 구경해봐야겠습니다.
벡터빌더 | Revolutionize Gene Delivery from Research to Therapy
Prevalence of errors in lab-made plasmids across the globe Bai, et al. BioRxiv. 2024 Jun. This study raises alarm on the quality of lab-made vectors by showing that nearly 50% of them have design or sequence errors.
www.vectorbuilder.kr
읽고있는 논문에선 앞서말한 dox 주입 후 inducible OSKM mice(발현을 확인할 genes가 여러 개)의 intestine에서 2A peptide를 immunofluorscence(IF) 방법으로 염색해 잘 들어갔는지 확인했습니다. Tet-On system이니 dox를 주입했다면 유전자 발현이 활성화되었어야 했을테니까요.
기본적인 내용이긴 한데, 기본이 가장 중요한 만큼 염색법이 나온 김에 다음 글에서는 염색 방법인 'Immunohistochemistry(HIC) & Immunofluorescence(IF)를 정리해보겠습니다. 또한, 생물학 연구에서 중요한 기본 과정인 Cell fixation에 대해서도 정리해보겠습니다.
참고자료
0) 약학정보원, 약물백과, 테트라사이클린(tetracycline) & 독시사이클린(doxycycline)
1) Das AT, Tenenbaum L, Berkhout B. Tet-On Systems For Doxycycline-inducible Gene Expression. Curr Gene Ther. 16(3):156-67. (2016).
2) genoway, TET System: Controlled gene expression, URL : https://www.genoway.com/technologies/tet-system
3) Melina Fan, Plasmids 101: Multicistronic Vectors addgene Blog, 2014, URL : https://blog.addgene.org/plasmids-101-multicistronic-vectors
4) Martinez-Salas Encarnacion , Francisco-Velilla Rosario , Fernandez- Chamorro Javier , Embarek Azman M. Insights into Structural and Mechanistic Features of Viral IRES Elements. Frontiers in Microbiology. 8. 2018
5) Liu, Z., Chen, O., Wall, J.B.J. et al. Systematic comparison of 2A peptides for cloning multi-genes in a polycistronic vector. Sci Rep 7, 2193 (2017).
6) SnapGene, Cloning PCR Products, URL : https://www.snapgene.com/guides/cloning-pcr-products
7) BD Biosciences, Immunohistochemistry, IHC 원리 URL : https://www.bdbiosciences.com/ko-kr/learn/applications/ immunohistochemistry?tab=Overview




