안녕하세요, 이번 글에선 저번 챕터 1에 이어 '생물정보학 입문' 책 챕터 2를 정리해보고자 합니다.
[41일차] 생물정보학 입문 책 정리 Ch. 01 :: 서론
안녕하세요 이번 글에선 최근에 공부하고 있는 '생물정보학 입문 제 2판' 책 첫 챕터를 정리해보고자 합니다. 본 내용에는 오류가 있을 가능성이 있으며, 자세하고 정확한 내용은 본 책 1)을 참고
tkmstudy.tistory.com
과학의 발전으로 우리는 우리의 형질을 결정하는 유전자가 염색체에 존재하고, 각 유전자가 DNA 서열에 해당한다는 것을 알게 되었습니다. 결국 우리가 우리와 같은 외모를 갖게 되고, 특정 질병에 취약하게 되고, 그럼에도 생존해 나갈 수 있는 건 바로 우리 몸 안에 소중하게 보관된 DNA가 우리의 형질을 결정하기 때문일 수 있습니다. 결국 우리는 우리의 형질이 잘못된 방향으로 흘러가지 않도록, 우리의 형질 결정에 영향을 미치는 DNA를 이해해야 할 것입니다.

이러한 일환으로 우리 몸의 DNA 서열을 해독하여 유전자 지도로 나타내고자 하는 '인간 게놈 지도'를 완성하고자 하는 노력이 이어져왔는데요. 대표적인 프로젝트가 20년 전에 진행되었던 '인간 게놈 프로젝트'였죠. 당시 절반 이상이 규명되지 못한 Y 염색체(염기서열 반복부위가 많기 때문)의 DNA 서열이 최근 롱리드 시퀀싱 기술을 통해 해독되어 인간 게놈 지도의 마지막 퍼즐이 완성되었습니다 2). 그렇지만, 환상적인 요리 레시피 한 줄 한 줄을 모두 다 안다고 해서 반드시 환상적인 요리를 만들 수 있는 건 아니듯이 DNA 서열을 모두 안다고 해서 우리 몸이 어떻게 기능하는지 완벽하게 이해할 수 있는 것은 아닙니다.
게놈 서열의 완성은 전체 매핑 작업의 정점이다. 그러나 게놈 서열은 유기체의 유전 정보를 일차원적이고 정적인 형태로만 기술한다는 점을 다시 한번 강조할 필요가 있다. 게놈 지도는 이 정보가 시공간에서 어떻게 구현되는지, 순차적 발달 프로그램에 의해서 유전자 발현이 어떻게 구성되는지, 그리고 게놈 자체를 넘어 유전자 발현에 대한 제어를 포함하여 유기체의 구조와 활성이 환경과 경험 및 후생유전학에 미치는 영향에 대한 정보를 우리에게 제시하지 못한다. 1)
가장 간단한 예로, 유사한 유전 정보를 가진 쌍둥이가 다른 환경에서 자라게 되면, 사뭇 다른 표현형을 가지게 되는 사례가 있죠. 그만큼 우리의 형질(표현형)은 단순히 DNA 서열로만 결정되기보단 그 DNA 서열에 있는 유전자의 발현을 조절하는 여러 후생유전학적 신호들과 환경적 요인들에 영향을 받는다고 할 수 있겠습니다. 작년에 후생유전학에 대한 책을 읽었던 기억이 나네요.
[16일차] 장연규 연세대 시스템생물학과 교수님의 후성유전학 관련 책 리뷰, < 유전자 스위치 >
안녕하세요, 오늘 글에서는 최근에 재밌게 읽은 과학책인 장연규 연세대 시스템생물학과 교수님이 집필한 '유전자 스위치'에 대해 간단하게 리뷰해보고자 합니다. 참고로 책 속 내용 정리에 있
tkmstudy.tistory.com
DNA 서열 자체도 같은 종의 사람이라 해도 조금씩 다릅니다. 다시 말해, 우리는 개인마다 고유한 DNA 서열을 가지고 있습니다. DNA를 통해 범인을 찾아내고, 친자 확인을 할 수 있는 이유가 바로 여기에 있죠. 그렇다면 어떤 DNA 서열이 개인마다 어떻게 다른걸까요? 다르다고 해도 우리가 다르다는 걸 확인하고 그것이 특정 개인의 DNA임을 확인할 수 있어야 범인을 식별하고 친자를 확인할 수 있을 텐데, 이는 어떤 생명공학 기술을 활용해야 가능한 걸까요? 먼저 사람마다 다른 DNA 서열의 특징부터 살펴보겠습니다.
우리의 게놈에서는 특정한 짧은 염기서열이 동일한 염기서열로서 반복되는 특성이 있습니다. 이러한 짧은 염기서열들은 게놈에서 한번 만 나타날 수도 있고, 서로 다른 염색체에서 서로 다른 길이로 여러번 나타날 수 있습니다. 이때, 그 특정 짧은 염기서열이 반복되는 길이는 개인마다 고유하게 다르며, 이 특징은 가족에게 유전되는 특성이 있다고 합니다. 그래서 친자 확인에 DNA를 활용할 수 있는 것입니다.
그럼 이제 친자 확인 및 형사 사건에 사용되어온 우리에게서 고유하게 존재하는 '짧은 길이의 반복되는 유전자 서열'에 대해 알아보겠습니다. 그것은 바로, 10~100 bp 길이의 영역을 포함하는 미니위성(ministalite)라고 불리는 'Variable number tandem repeats(VNTR)'입니다. 물론, 요즘은 다른 짧은 반복 서열을 주로 사용하고 있다고 하는데요. 그것은 바로 2~5bp를 포함하고 있는 짧은 텐덤 반복(STR)인 미소위성(microsatelite)이라 불리는 'Short Tandem Repeat Polymorphisms(STRP)'이라고 합니다. STR는 일반적으로 10~30번 연속적으로 반복된다고 하죠.
STRP는 VNTR에 비해 표지자로서 몇 가지 장점이 있는데, 그 중 하나는 인간 게놈에서 더 균일하게 분포되어 있다는 것이다. 이러한 표지자는 발현된 유전자 내에 위치할 필요가 없으며, 종종 발현된 유전자 내에 위치하지 않는다. 헌팅턴병 및 특정 다른 질병 유전자에 존재하는 CAG 반복은 예외이다. 1)
STR이 인간 게놈에게서 더 균일하게 분포한 것이 개인 식별에 장점이 되는 이유는 비슷한 위치에서 있어 함께 유전되는 특징인 'linkage'에 의한 영향을 최소화할 수 있기에 개인 간 다양성이 높기 때문으로 추측됩니다. 이때, 하나의 STR로만 개인 간의 STRP를 특징짓는 건 불가합니다. 왜냐하면, STR 하나의 길이의 가변성이 전체 인구를 구별하기에 충분하지 않기 때문입니다. 앞서 STR이 일반적으로 10~30번 연속적으로 반복한다고 했는데 우리 전체의 인구가 약 70억인데 하나의 STR로 사람을 구별하긴 어렵겠죠.
따라서 보통 여러 STR의 조합과 성별 구분(XX or XY)으로 개인을 식별한다고 하는데요. 저자가 말하길, 영국 국립 데이터베이스는 양쪽 가닥의 직렬 반복수를 10개의 STR로 지정하고(두 개의 대립유전자를 받으므로, 각 STR에 대해 두 개의 숫자가 기록되어 2 x 10), 값이 XX 또는 XY인 아멜로게닌 테스트를 사용하는 SGM+ 프로토콜을 사용한다 1)고 합니다. 미국의 경우엔 13개의 STR과 성별을 보고하는 아멜로게닌이 포함된다고 합니다. 인구가 더 많아서 그런가 싶습니다. 암튼 그렇다면 성별을 확인할 때 왜 아멜로게닌 유전자를 포함하는걸까요? 그것은 바로, 인간의 아밀로게닌 유전자가 성염색체에서 구분되는 차이가 있기 때문입니다.
인간의 아밀로게닌 유전자는 X 및 Y 염색체 모두에 나타난다. X 복사본은 Y 버전에 비해 1에서 6 bp의 인트론 삭제가 나타난다. 기존 성별 시험에서는 적절한 프라이머를 사용하여 PCR을 통해 Y 염색체에서 112bp, X 염색체에서 106bp 조각을 증폭한다. 젤 상의 2개의 밴드는 남성 DNA 원본을 의미하고, 1개의 밴드는 여성을 의미한다. 돌연변이가 이 시험을 무효화하는 경우, 다른 Y 염색체 표지자를 사용할 수 있다. 1)
돌연변이의 예로 Y 염색체의 아밀로게닌 부위에 돌연변이가 생겨 프라이머 결합이 어려운 상황이 있을 수 있겠습니다. 그렇게 되면 남자임에도 Y 밴드(112bp)가 전혀 증폭되지 않거나, 예상보다 더 작거나 큰 DNA 조각을 만들게 될 수 있죠. 결국, 남자를 여자로 오판하게 될 수 있습니다. 이럴 땐 위의 구절에서처럼 다른 Y 염색체 표지자를 사용한다고 합니다.
이러한 방법으로 개인을 식별할 때는, 사건 현장에서 확보한 DNA와 용의자의 DNA 간에 여러 STR 조합의 반복 길이를 비교하고 아멜로게닌 유전자를 분석해 성별을 확인함으로써 범인을 특정할 수 있습니다. 또한 친자 확인 시에는 STRP 마커에서 자녀의 대립유전자가 부모의 것과 일치하는지 살펴보면 되는데요. 이때 자녀는 어머니와 아버지 양쪽으로부터 각각 상염색체 STR을 물려받으며, 각 부모의 염색체 쌍 중 하나만을 물려받습니다. 따라서 부모의 STRP 중 절반이 자녀에게 나타나고, 자녀가 갖는 모든 STR는 반드시 어느 한 부모로부터 유래해야 한다고 볼 수 있겠습니다. 감수분열에서 부모 각각의 염색체에서 두개의 반수체 중 하나씩만 자녀에게 가기에 자녀에게 가지 않은 부모에게 남은 하나씩의 반수체는 자녀에게 유전되지 않으니까요. 당연히 이는 반수체 각각이 STR 반복 길이가 고유하게 다를 수 있다는 걸 전제로 합니다.
이제 STR의 반복 길이로 개인을 식별해 범인 추적 및 친자 확인을 할 수 있다는 것을 알게 되었습니다. 그렇다면 개인의 고유한 STR 반복 길이는 어떻게 확인할 수 있을까요? 이 방법은 간단합니다. 먼저 STR 구간 양쪽을 에워싸는 특이적 프라이머를 사용하여 PCR을 수행하면, 각 STR 구간에서 반복 횟수에 따라 다양한 길이의 DNA 조각들이 생성됩니다. 이 DNA 조각들은 전기영동을 통해 피크 형태의 프로파일로 분석되며, 그 피크의 길이를 비교하여 각 STR 위치별 개체 간의 길이 차이를 확인할 수 있습니다.
이제 DNA 서열 결정 기술의 역사를 살펴보겠습니다.
과거에는 컴퓨터 기술이 발달하지 않아 Illumina 시퀀싱과 같이 수많은 리드의 염기서열을 병렬로 분석하는 기술은 존재하지 않았습니다. 이러한 기술이 개발될 수 있었던 것은 DNA 서열을 규명하고자 하는 과학자들의 끊임없는 노력 덕분입니다. 이러한 선구자 중 한 명이 바로 케임브리지의 생화학자 Frederick Sanger입니다. 아래 영상은 아직 안봤는데 나중에 보려고 링크를 아래 해두었습니다.
Frederick Sanger는 초기 DNA 중합효소를 활용하여 DNA 서열을 결정하는 혁신적인 방법을 개발했습니다. 이 방법은 DNA 복제 과정에서 DNA 중합효소가 프라이머에 상보적인 뉴클레오티드를 추가하며 DNA 가닥을 확장하는 원리를 기반으로 합니다. Sanger 방법에서는 디데옥시뉴클레오시드 삼인산(ddNTP)을 사용하여 확장을 중단시킵니다. 이로 인해, 각기 다른 뉴클레오티드로 끝나는 일련의 중첩된 DNA 조각들이 생성되고, 젤 전기영동과 형광 크로마토그램을 통해 이 조각들의 길이와 종료 뉴클레오티드를 확인함으로써 최종적으로 DNA 서열을 읽어낼 수 있습니다.
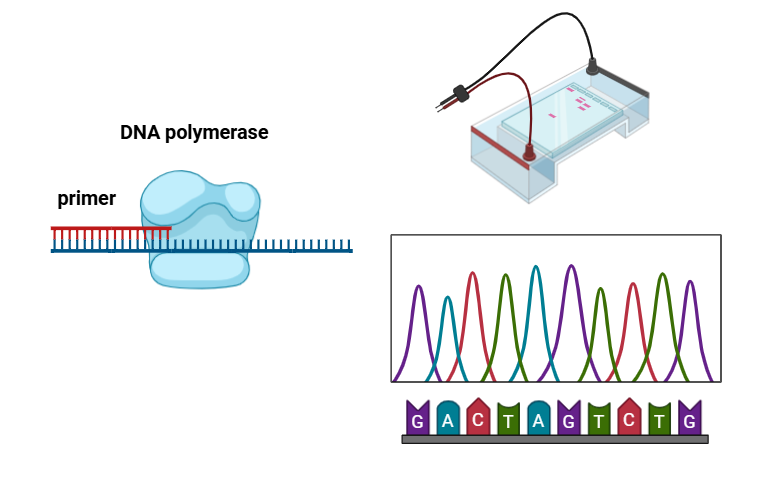
초창기에는 방사성 동위원소를 이용해 끝에 위치한 뉴클레오티드를 파악했다고 하는데요. 이는 건강에 해롭고 비용도 만들기에 형광 염료를 리포터로 사용하는 방식으로 전환되었다고 합니다.
네 가지 디데옥시뉴클레오시드 삼인산에 서로 다른 형광 염료를 부착하면, 각 조각이 연장을 중단시킨 디데옥시뉴클레오티드에 따라 다른 신호를 생성한다. 네가지 반응은 '같은 포트에서' 수행될 수 있으며, 전기영동은 이를 단일 레인(single lane)에서 분리한다. 고정된 지점에 집중된 레이저는 각 조각이 지날 때마다 그것들을 식별한다. 결과는 서열의 연속된 염기에 해당하는 연속된 정점(peak)으로 표시될 수 있다. 1)
다시 한번 정리하자면, 네 종류의 형광이 붙은 디데옥시뉴클레오타이드를 한 번에 반응시키면, DNA 합성 과정을 거쳐 특정 염기에서 종결된(형광이 붙은 디데옥시뉴클레오티드는 3' 탄소에 수산기(-OH)가 없어 DNA 사슬 연장을 멈추게 하기 때문) 다양한 길이의 조각들을 생성합니다. 이 조각들을 전기영동으로 크기별로 분리한 뒤, 레이저로 형광 신호를 감지하면 중첩된 크기별로 나열되어 있을테니까 어떤 염기가 순차적으로 붙어있는지 알게 되고, 이를 통해 전체 DNA 염기서열을 읽어낼 수 있게 됩니다. 이때, 형광 신호 감지에는 각 염기마다 피크로 나타나는 형광 크로마토그램 형태로 확인하게 되고, 연속된 피크들이 DNA 서열 정보를 나타낸다고 볼 수 있겠습니다.
이 과정에서 오류의 가능성을 배제하긴 어려울 것입니다. 어떤 DNA의 특정 서열 위치에서 오류가 발생하거나 신호가 잘못 검출되어 일반적인 염기와 다른 염기가 나타날 수 있기 때문입니다. 그만큼 서열 결정의 품질 측정 지표로서, 서열 결정의 정확도를 측정하는 'Phred 점수 q'를 활용하는데요. 이는 보고된 염기가 정확할 확률을 보여줍니다. 염기가 오류일 확률이 'p'라면, Phred 점수 q = -10 log10p가 됩니다. 본 기준에 따라 오류 확률이 0.1(10개 중 염기 1개가 잘못됨)라면 점수 q는 10이고, 0.001(1000개 염기 중 염기 1개가 잘못됨)이라면 점수 q는 30이라고 계산할 수 있겠습니다.
이후 시간이 흘러 DNA 염기서열 분석 기술은 과학과 기술의 발전 속도에 맞춰 급격한 발전을 이루게 됩니다. 그 중심점에는 'NGS(Next Generation Sequencing)' 기술의 등장을 빼놓을 수 없을 것입니다. 우리는 NGS 방법을 통해 보다 'High Throughput'으로 신속하고 저렴하게 염기서열을 분석할 수 있게 되었습니다. 가장 놀라운 혁신은 여러 개의 DNA 샘플을 동시에 시퀀싱할 수 있게 된 점이 아닐까 싶습니다.
새로운 기술은 긴 분자의 매핑된 조각들을 분리하고 서열을 결정하는 전략에서 ‘샷건 서열 결정(shotgun sequencing)’으로 전환할 수 있게 했다. DNA는 작은 조각으로 잘려지고, 각 조각에서 수집된 서열 정보는 전체적으로 일관된 서열로 조립된다. 1)
즉, 샷건 시퀀싱은 DNA 전체를 말 그대로 샷건처럼 작은 조각으로 무작위로 잘라낸 후 각 조각을 개별적으로 해독(시퀀싱)한 뒤, 해독된 조각들을 컴퓨터를 이용해 재조합하여 원래의 전체 DNA 서열을 복원하는 기술입니다. 그냥 한번에 긴 조각을 해독하는 게 더 좋지 않을까 싶기도 한데, 여기엔 긴 조각을 읽지 못하는 기술적인 한계가 있고, 긴 조각을 읽는 것보다 작은 조각을 조립해서 전체 서열을 추론하는 게 더 효율적인 이유도 있다고 볼 수 있겠습니다. 물론, 변이를 확인하거나 반복이 많아 까다로운 부분의 DNA 서열을 해독하는 데에는 긴 조각 해독이 필요하기에 그에 맞는 롱리드 시퀀싱 기술도 나중에 등장하긴 했습니다. 그에 대해선 이후에 소개하도록 하겠습니다.

각각의 잘라진 조각을 맞추는 건 퍼즐 맞추기에 빗대어 설명할 수 있지 않을까 싶습니다. 우리는 퍼즐을 맞출 때 퍼즐의 모양, 그리고 퍼즐에 그려진 그림을 보고 퍼즐들을 순차적으로 조립해나갑니다. 잘라진 DNA 조각 같은 경우엔 일반적인 퍼즐과 달리 중첩된 영역이 있기에(ex. AT'ACG', 'ACG'AG) 겹치는 부분을 고려하여 연속적으로 이어 맞추면 됩니다.
참조 서열(reference genome)이 있는 경우엔 더 간단합니다. 퍼즐의 정답 이미지가 제공되는 꼴이니까요. 그 위치에 맞게 조각난, 그러면서도 심지어 중첩된 퍼즐들을 맞추어나가면 됩니다(물론 각 퍼즐들의 구성 요소가 A T G C 네 가지 염기로만 차이가 나기에 헷갈리긴 하지만요). 우리는 인간 게놈 프로젝트를 통해 퍼즐의 정답에 가까운 이미지를 만들어 냈고, 이제 특정 DNA 샘플의 서열 해독에 있어서 참조 서열에 매핑해 보다 정확하고 신속하게, 그리고 효율적으로 서열 조립 및 결정을 할 수 있게 되었습니다. 참고로 참조 서열을 사용하지 않고 전체 게놈 서열을 결정하는 건 'De novo sequencing'이라고 합니다. 결국, De novo sequencing을 통해 Resequencing(참조게놈서열이 알려진 종의 개체 서열을 결정하는 것 1))을 할 수 있는 '인간 게놈 지도'를 완성할 수 있었던 것으로 볼 수 있겠습니다.
그러나, 다음과 같이 잘라진 DNA 조각이 반복 서열 안에 속하는 서열이라 위치를 특정 짓기 어려운 경우엔 어떻게 해야할까요? 이 경우엔 퍼즐의 정답 이미지 즉, 참조 서열이 있다고 해도 해당 조각을 어디에다가 끼워 넣을지 막막합니다.

참고로, 여기서 'CAGCAGCAG.. '로 되어있는 DNA 조각은 서열이 한번 읽혔다고 해서 '리드(read)'라고 합니다. 그리고 이와 같이 '조각의 한쪽 끝에서만 서열이 보고되는 기술 1)'을 'Single-end read'라고 합니다. 그렇다면 왜 조각 전체 서열을 보고하지 조각의 한쪽 끝에서만 서열을 보고하는걸까요? 이 또한 앞서 말했듯 기술적 한계라고 볼 수 있겠습니다. 그말은 즉슨, 기술의 발전이 과학의 발전으로 이어질 수 있다는 말이기도 하겠죠. 생물정보학의 발전은 이러한 흐름으로 이어지고 있는 듯합니다. 다시 질문으로 돌아와서 CAGCAGCAG 서열(..도 CAG 반복으로 총 50 bp 정도라고 가정, 참조 서열의 CAG 반복은 30이라고 가정(30x3 = 90))은 어디에 끼워넣을 수 있을까요? 이미지에서 오른쪽에서부터 첫번째 C에서 시작해도 될 것 같고, 네번째 C에서 시작해도 문제가 없을 듯 합니다(참고로, 헌팅턴 증후군의 경우 CAG 서열이 약 30개 정도 반복되는 특징이 나타납니다. 그래서 예시로 한번 대충 만들어봤습니다).
이처럼 반복 영역 길게 있을 경우 리드들을 조합해, 콘티그(연속된 리드들 조립한 것)를 만들고, 콘티그로부터 완전한 서열을 얻긴 쉽지 않을 것입니다. 이러한 문제를 해결하는데 도움이 되는 기술이자, 1+1으로 서열을 해독하는 것이 DNA 조각의 양쪽 끝에서 서열을 보고하는 기술인 'Paired-end read'입니다.
Paired-end read는 양 끝에서 약 50bp 정도 떨어진 부분까지 서열을 결정할 수 있으며, 서열이 결정된 영역 간의 거리는 대략적으로만 알려져 있다고 합니다. 그렇지만 그렇게 읽힌 Paired-end read들을 참조 서열에서 조립해나가면 보다 효과적으로 전체 서열을 결정할 수 있습니다. 그리고 앞선 반복에 따른 위치 결정 문제도 해결할 수 있습니다. 다음 이미지를 보면 됩니다.

즉, 왼쪽 단편 끝 부분의 서열이 보고된 부분은 반복이라 어느 위치에 끼워넣어야 할지 애매하지만, 오른쪽 단편 끝 부분은 특이적이기에 결국 해당 리드의 참조 서열 내 위치를 파악할 수 있게 되는 것입니다. 이에 더해, 우리는 하나의 리드로부터 두 개의 연속된 약 50bp 정도의 서열을 한번에 파악할 수 있게 되었습니다. 이러한 장점으로 최근에 대부분의 시퀀싱 기술에 Paired-end read를 활용한다고 하네요.
조립의 후반 단계에서, 페어드-엔드 리드는 콘티그가 어떻게 정렬될 수 있는지를 나타낼 수 있다. 상당한 콘티그가 결정되었지만, 갭으로 인해 콘티그의 순서가 불확실해지는 조립단계를 생각해보면, 서로 다른 콘티그를 연결하는 페어드-엔드 조각은 콘티그를 통합된 스캐폴드로 결합시킬 수 있다. 1)
물론, Paired-end read 기술이 개발되었다고 해도 DNA 염기 서열을 결정하는 건 쉬운 일이 아닙니다. 엄청난 길이의 DNA 서열에서 Paired-end read는 아주 적은 정보를 가지고 서열 내 위치를 추론하는 것이고, 무엇보다 DNA 서열 내 변이와 Paired-end read보다 긴 반복은 본 기술이 커버하기 어려울 수 있기 때문이죠.
이번엔 DNA 서열 재구성에 있어서 컴퓨터적 측면을 살펴보도록 하겠습니다. 서열에 반복이나 오류가 없다면 리드들의 조립해 서열을 재구성하는 것은 간단한 문제일 수 있습니다. 중첩된 영역들끼리 연결하면 끝이니까요. 중첩된 영역의 연결은 서픽스 트리를 통해 컴퓨터적인 측면에서 수행할 수 있습니다. 다음은 책 속 예시인 'inning'라는 단어에 대한 서픽스 트리를 나타낸 것입니다. 여기서 'inning'의 서픽스는 'inning', 'nning', 'ning', 'ing', 'ng', 'g'가 될 수 있고 본 서픽스 트리는 루트 노드에서부터 내려가며 모든 서픽스들을 표현하고 있습니다.

두 리드의 중첩된 영역 식별 즉, 한 리드의 접미사와 다른 리드의 접두사 간의 일치를 식별은 서픽스트리에 '구분 문자'를 나타냄으로써 가능하게 만들 수 있습니다. 예로, 간단하게 'gaccgc'와 'ccggac'라는 2개의 리드가 있다고 가정해봅시다. 여기서 첫번째 문자열엔 구분문자 '#'를 추가하고 두번째 문자열에 구분문자 '$'를 추가하면 결합된 문자열은 'gaccgc#ccggac$'이 됩니다. 이를 서픽스트리로 나타내면 다음과 같습니다(챗GPT가 알려준 파이썬 코드로 구현해서 얻은 이미지입니다).

그림에서 ‘gac’ 경로를 살펴보면(맨 오른쪽), 그 아래에 첫 번째 문자열에서 ‘gac’로 시작하는 리프와 두 번째 문자열에서 ‘gac’로 끝나는 리프가 동시에 존재하는 것을 확인할 수 있습니다. 이를 통해 두 문자열이 ‘gac’라는 공통 부분 문자열을 공유한다는 사실을 알 수 있죠. 언뜻 보면 ‘gaccgc’와 ‘ccggac’의 일치 구간에서 ‘gac’가 바로 드러나니, 단순 비교로도 금방 찾아낼 수 있을 것처럼 보이지만, 실제로는 더 긴 구간이나 여러 길이로 겹치는 부분들을 모두 고려하여 서열을 재구성해야 합니다. 이 때문에, 서픽스 트리를 직접 그려가면서 이러한 겹치는 조각들을 단계적으로 조립해 나가는 방법이 유용하다고 합니다.
그렇지만 서픽스트리는 그 서열이 맞다는 가정하에 조립해야 한다는 한계가 있습니다. 서열에 반복과 오류가 있어서 조각들의 순서가 고유하지 않을 때 우리는 또 다른 '그래프'를 통해 조립 중의 모호성을 나타낼 필요가 있습니다. 이러한 모호성을 그래프로 표현하는 대표적인 방법이 바로 '중첩 그래프'와 '드 브루인 그래프'라고 합니다.

먼저 중첩 그래프는 위와 같이 '노드'를 '리드'로 나타내고, 겹치는 리드를 엣지로 연결하는 형태를 띠는데요. 이때 엣지는 5개 이상의 염기의 중첩을 의미한다고 합니다. 참고로 위의 이미지는 책의 이미지를 그려본겁니다. 여기서 곡선 화살표는 '전이적 중첩(transitive ovelaps)'을 의미한다고 합니다. 그렇게 R1과 R3, R2와 R4도 겹침을 표현합니다. 이후 R4의 리드 말단 서열이 R5와 R8 리드의 초기서열 둘 다와 일치합니다. 그렇게 갈라짐이 발생하게 됩니다. 그래프가 그려지고 나면, 경로를 이어 리드들을 조립합니다.
리드와 일치하는 조립을 찾으려면 중첩 그래프를 통해 그래프를 따라가면서 각 노드를 정확하게 한 번 방문하는 경로를 찾아야 한다.
'각 노드를 정확하게 한번 방문하는 경로'라는 점에서 이는 '해밀턴 경로'라고 할 수 있겠습니다. 해밀턴 경로는 '단순 그래프에서 각 정점을 정확히 한 번씩만 통과하는 경로 4)'를 말합니다. 즉, 모든 꼭짓점을 한번씩 만 지나면서 한 점당 두 개의 선(들어가는 간선과 나가는 간선)만 지나는 그런 경로라고 볼 수 있겠습니다.
두번째 그래프인 '드 브루인 그래프'는 알려진 조각들을 동일한 길이의 영역으로 세분화합니다. 즉, 리드를 3-bp의 하위 조각(3-mer)으로 세분화합니다.
드 브루인 그래프에서 노드는 리드에서 나온 연속된 3개의 염기서열 조각이다. 엣지는 2개의 염기 중첩에 해당한다. 상류 노드를 레이블링하는 삼중항(triplet)의 2-3 염기가 하류 노드를 레이블링하는 삼중항의 1-2 염기와 중첩된다. 1)
드 브루인 그래프는 다음과 같이 그려볼 수 있습니다.

드 브루인 그래프에서 리드와 일치하는 조립을 찾으려면 그래프를 통과하는 모든 엣지를 정확하게 한번 씩 방문하는 경로를 찾아야 합니다. 이러한 경로를 '오일러 경로(Eulerian path)'라고 합니다. 앞서 해밀턴 경로는 노드를 정확하게 한번씩 방문해야 했죠.
해밀턴 경로는 모든 엣지를 통과하지 않을 수 있지만, 오일러 경로는 반드시 모든 엣지를 통과해야 한다. 오일러 경로는 하나 이상의 정점을 여러 번 통과할 수 있지만, 해밀턴 경로는 그렇게 할 수 없다. 계산적으로, 해밀턴 경로를 찾는 것은 오일러 경로를 찾는 것보다 훨씬 어렵다. 1)
생각해보니 작년 이맘 때 쯤에 '생물정보학 알고리듬' 책 앞부분을 잠깐 읽으면서 해밀턴 경로와 오일러 경로를 공부했던 기억이 나네요.
[생물정보학 알고리듬 3장] '리드(read)'로부터 유전체 뉴클레오티드 서열 조립하기 :: 드 브루인
안녕하세요 TKM입니다! 그동안 영어 공부겸 '생물정보학 알고리듬' 책 내용을 영어로 한 챕터씩 정리해왔는데 그러다보니 제가 제대로 내용을 쓰고 있는지, 그리고 제대로 공부가 되고 있는건지
tkmstudy.tistory.com
다음 영상에서 해밀턴 경로와 오일러 경로의 차이에 대해서 잘 설명해주니 참고 바랍니다!
여기까지 챕터 2를 정리하고, 또 공부하다가 정리할 게 있으면 찾아오겠습니다. 감사합니다!

참고자료
1) 저자 ARTHUR M. LESK. 번역 이용석,강병철,김익수 외 3명, 생물정보학 입문 제2판, 월드사이언스, 2024
2) 최인준 기자, "Y염색체 완전 해독… 인간 게놈지도 20년만에 마지막 퍼즐 맞춰", 조선일보, 2023, URL : https://www.chosun .com/economy/science/2023/08/31/4CWZPONOORCSFJVMTIVKP6SI7A/
3) 제한효소, 미생물학백과, URL : https://terms.naver.com/entry.naver?docId=5894393&cid=61232&categoryId=61232
4) 해밀턴 경로, NAVER 국어사전, URL : https://ko.dict.naver.com/#/entry/koko/35989366c2214a96b8330a2a01359ce0



