이번 글에서는 scRNA data를 기반으로 R 환경 Seurat 패키지를 통해 개별세포분석을 해보고자 합니다.

이를 위한 data로는 전에 수강을 완료한 KOBIC 교육센터의 '예제 데이터를 활용한 단일세포 전사체 데이터 분석' 영상에서 무료로 제공한 예시 데이터를 활용하였습니다.
참고로 예시 데이터는 10X GENOMICS에 있는 '10k Peripheral Blood Mononuclear Cells (PBMCs) from a Healthy Donor, Single Indexed'를 100,000줄만 가져왔다고 하며, 해당 세포는 single cell로 연구가 많이 되어 있어서 toy set으로 잘 활용되고 있다고 합니다.
10k Peripheral Blood Mononuclear Cells (PBMCs) from a Healthy Donor, Single Indexed - 10x Genomics
10k Peripheral Blood Mononuclear Cells (PBMCs) from a Healthy Donor, Single Indexed Single Cell Gene Expression dataset analyzed using Cell Ranger 4.0.0
www.10xgenomics.com
본 데이터 활용을 희망하시는 분들은 INCOEDU의 KOBIC 교육센터에 들어가보시길 추천드립니다.
차세대 생명정보 온라인 교육 | KOBIC 교육센터
KOBIC 차세대 생명정보 교육은 바이오 데이터 분석 및 활용을 위한 IT 기술(프로그래밍 언어, 리눅스)과 바이오 데이터 분석 전문기술을 제공합니다.
edu.insilicogen.com
본 강의에서 제공한 예시 데이터는 filtered_feature_bc_matrix 파일로 그 안에는 'barcodes.tsv', 'features.tsv', 'matrix.mtx'라는 파일이 존재합니다. 본 파일에 대한 설명은 다음과 같습니다.

여기서 barcodes.tsv는 각 유전자가 어떤 세포에서 유래된 시퀀스인지 알려주는 Cell ID를 담은 파일, features.tsv는 각 유전자 정보를 의미하는 Gene ID (다른 antibody를 담은 경우 feature 정보)를 담은 파일, matrix.mtx는 유전자의 발현량 정보를 담은 파일로, '33509 1 60'이면 33509번째 유전자가 있는 1번 세포로 read count는 총 60개 라고 볼 수 있습니다.
이러한 세 파일을 R 환경인 R studio로 가져왔는데, 여기서부터 전 글에서 소개했던 BRIC scRNA-seq data 분석법 글을 따라하면서 연습해봤습니다.
[당신의 논문 동료] scRNA-seq data 분석법 - R 설치부터 Seurat까지 & Public scRNA-seq 분석 사이트 소개 |
1. 들어가며 대학교 3학년 연구생 인턴을 하던 시절, 한창 DNA 클로닝을 배우고 있을 때 선배께서 “손을 잡고 가르쳐 주는...
www.ibric.org
[당신의 논문 동료] scRNA-seq data 분석법 – Plot 4 종류부터 DEG까지 | 뉴스 > Bio뉴스 > 동향 | BRIC
1. 들어가며 R을 활용하면서 가장 많이 나오는 에러에 대해 짚고 넘어가고 합니다. 1.1. R은 대소문자를 매우 엄격히 구분...
www.ibric.org
우선 제가 R은 이미 설치를 해둔지라 R studio를 켜고 세 가지 파일을 디렉토리에 가져왔습니다.
참고로, 분석법을 따라하고자 하시는 분들은 제 글을 참고하시기보다 위에 링크 글을 참고하시길 추천드립니다.제가 본 글을 쓰는 이유는 따라하면서 정리해보고자 함과 더불어 작심삼주 오블완 챌린지에 참여하기 위함이기에 대충 넘어가는 부분과 잘못 따라하는 부분이 있을 수 있습니다.
암튼 이렇게 세 개의 파일을 가지고 온 후 setwd( )나 'Set as working directory'를 활용해 'C:/'저장파일이름''으로 디렉토리를 지정해줍니다. 다음으로 분석에 필요한 패키지를 불러오기 위해 Seurat로 들어가서 Install 코드를 따와서 R studio에 붙여줍니다.
Tools for Single Cell Genomics
A toolkit for quality control, analysis, and exploration of single cell RNA sequencing data. Seurat aims to enable users to identify and interpret sources of heterogeneity from single cell transcriptomic measurements, and to integrate diverse types of sing
satijalab.org
저는 그냥 BRIC 글에 적힌 코드를 따라 적어 붙여넣어봤습니다.
remotes::install_github("satijalab/seurat", "seurat5", quiet = TRUE)
처음엔 이걸 하니까 Matrix 패키지가 업데이트 안되었다고 나와서 왜그런지 챗GPT한테 물어보니까 제가 R 버전을 옛날 꺼를 써서 그렇더군요. 그래서 최신 걸로 업데이트하고 다시 돌렸는데, 이번엔 Seurat 패키지가 없다길래 추가로 깔았습니다.
install.packages('Seurat')
그리고 글에 적힌 대로 필요한 패키지들을 불러왔습니다.
library(dplyr)
library(Seurat)
library(patchwork)
다음으로 pbmc.data라는 변수에 bacodes, features, matrix 파일을 담은 통합 파일인 'hg19(원래 파일이름은 filtered_feature_bc_matrix인데 글과 똑같이하려고 수정)'를 Read10X( )함수로 가져왔습니다.
pbmc.data <- Read10X(data.dir = "hg19/")
다음으로 입력 데이터 pbmc.data에 대해 'pbmc3k(3000개의 PBMC 데이터)'라는 프로젝트로 '세포 당 유전자 발현 수 최소 200개 이상', '특정 유전자의 최소 3개 세포에서의 발현'이라는 '필터 조건'을 지정해 Seurat 객체를 생성하고 pmbc라는 변수에 저장합니다.
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k", min.cells = 3, min.features = 200)
이제 기본적인 QC(quality control)을 진행할텐데 너무 많은 mRNA 정보가 담긴 세포(doublet 혹은 multiplet일 가능성 높음), 너무 적은 mRNA 정보가 담긴 세포(세포 또는 핵 찢어졌을 가능성 높음), 미토콘드리아 mRNA 비율 높은 세포(품질 낮은 세포 or 죽어가는 세포)를 필터링을 통해 제거해줄 것입니다.
어떤 기준으로 필터링을 진행할 것인지는 사용자 몫이라고 하는데, Plot을 그리면서 파악하면 된다고 하네요. 그 기준을 잡기 위해선 scRNA-seq 관련 논문의 method를 많이 참고하라는 팁을 주셨습니다.
먼저 세포에 mitochondrial gene이 얼마나 있는지 계산하기 위해 "MT-(사람의 경우, 생쥐는 mt-)"로 미토콘드리아 비율을 계산하는 함수를 만들어 그 값을 변수에 저장합니다.
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
이제 Violin plot으로 QC metrics를 만들어볼텐데, 이때 사용하는 함수는 VlnPlot( )입니다.
VlnPlot(pbmc, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)
그러면 다음과 같은 Plot이 등장합니다.
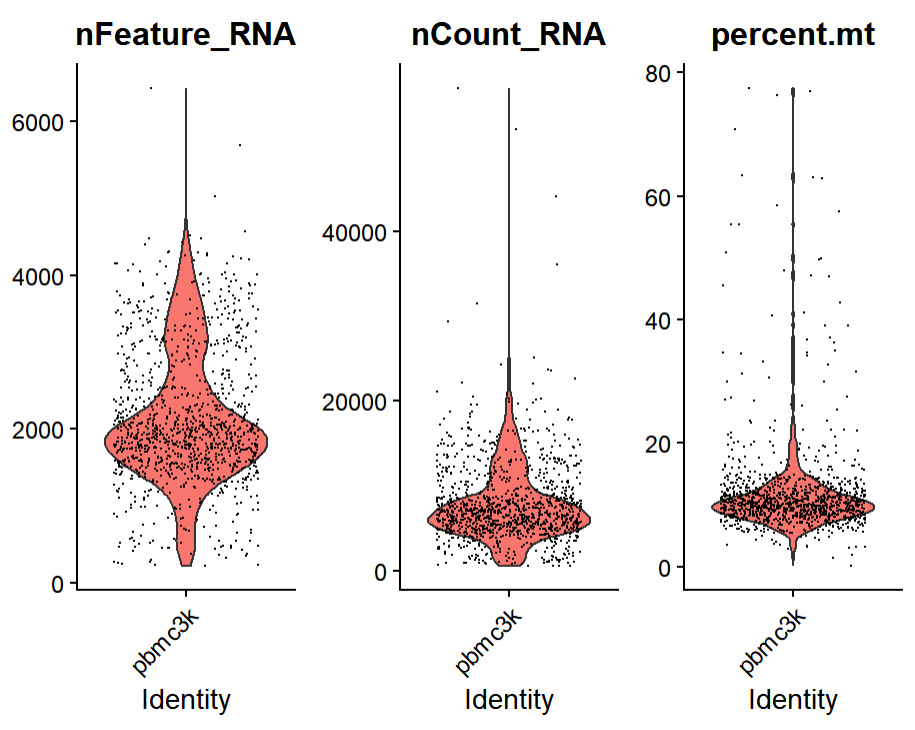
여기서 'nFeature_RNA'는 'feature' 즉 gene의 숫자를 의미하고, 하나의 세포에 몇 종류의 유전자 정보가 있는지 알려주는 plot이라고 합니다. plot을 보면, 2000 부근이 제일 많고, 4000 종류 이상의 유전자를 발현하는 세포는 doublet(하나의 droplet에 2개의 세포가 들어가 있는 경우)이라고 추론할 수 있겠습니다.
'nCount_RNA'는 'UMI count'를 의미하는데 UMI는 하나의 mRNA 당 붙는 tag이기에 UMI count는 mRNA 개수로 볼 수 있습니다. plot을 보면 50,000~10,000개 부근에서 데이터가 많아 보이고 20,000개 이상은 doublet으로 보입니다.
마지막으로 'percent.mt'의 경우 미토콘드리아에 코딩된 유전자의 비율로, 20% 이상은 outlier로 보입니다. 그러면 해당 기준에 따라 우선 nFeature_RNA 400 초과, 4500 미만, 미토콘드리아 비율 20 % 미만인 세포만을 선별(subset)하여 분석을 진행해보겠습니다.
pbmc <- subset(pbmc, subset = nFeature_RNA > 400 & nFeature_RNA < 4500 & percent.mt < 20 )
필터링한 데이터들에 대해 다시 violin plot으로 시각화해봤습니다.
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
VlnPlot(pbmc, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)

음.. nCount_RNA plot에 아직 outlier가 있는 것 같아서 괜히 신경쓰이니 이것도 nCount_RNA 25,000 미만으로 지정해 없애고 가겠습니다.
pbmc <- subset(pbmc, subset = nFeature_RNA > 400 & nFeature_RNA < 4500 & percent.mt < 20 & nCount_RNA < 25000 )
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
VlnPlot(pbmc, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)
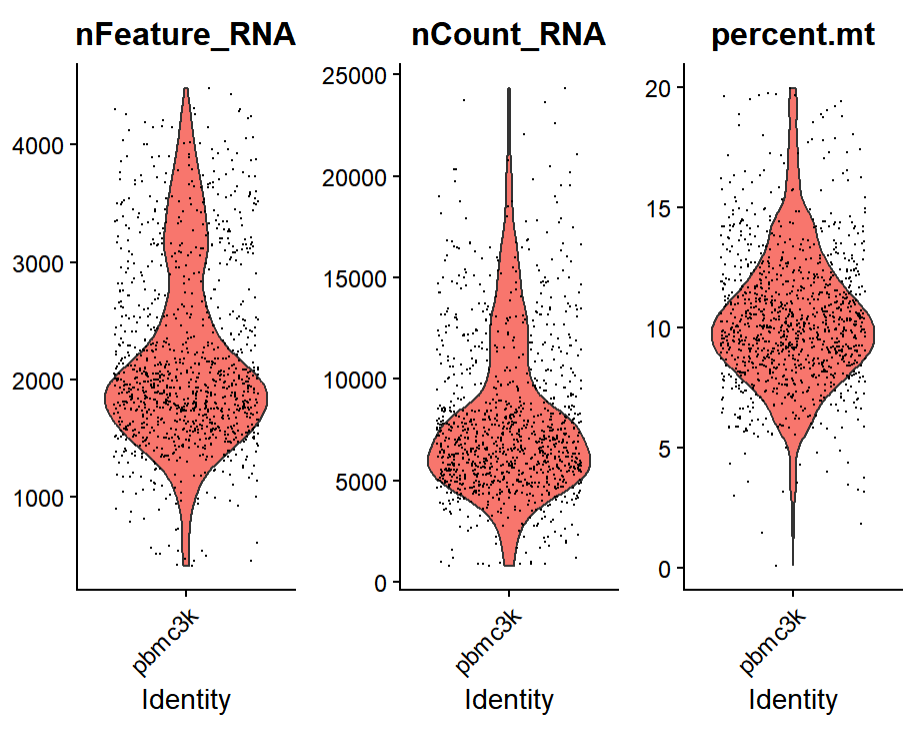
네 이제 뭔가 퀄리티가 좋은 세포들인 느낌이 듭니다. 물론 그렇지 않을 수도 있습니다. 그럼 다음 글에서 필터링한 해당 데이터를 가지고 Normalize부터 DimPlot까지 해보겠습니다. 지금 바로 하면서 글을 작성할 예정이지만, 작심삼주 오블완 챌린지에 7일차 참여로 치킨 응모를 하기 위해 오늘까지 4일차니, 5일, 6일, 7일 하루 하나씩 올려보도록 하겠습니다. 감사합니다!

참고자료
1) INCOEDU, KOBIC 교육센터, 예제 데이터를 활용한 단일세포 전사체 데이터 분석
2) 쿼카, [당신의 논문 동료] scRNA-seq data 분석법 – Plot 4 종류부터 DEG까지, BRIC
3) 쿼카, [당신의 논문 동료] scRNA-seq data 분석법 - R 설치부터 Seurat까지 & Public scRNA-seq 분석 사이트 소개, BRIC

'생물정보학(바이오인포매틱스)' 카테고리의 다른 글
| [24일차] BRIC의 scRNA-seq data 분석법 글 따라해보기 03 :: R 환경 Seurat의 DimPlot, FeaturePlot (1) | 2024.11.25 |
|---|---|
| [23일차] BRIC의 scRNA-seq data 분석법 글 따라해보기 02 :: Normalization부터 UMAP까지 (1) | 2024.11.24 |
| [22일차] 초보자를 위한 scRNA-seq data 분석법 글 추천, Edu labeling, Cre-loxp system (4) | 2024.11.23 |
| [22일차] 멘델의 정원, 생물정보학 기초, Fastq QC 강의 정리 (1) | 2024.11.23 |
| [21일차] KOBIC 교육강의 공부 04 :: 생물정보학의 미래 기술 (26) | 2024.11.17 |


