이번 글에선 저번 글에 이어 BRIC scRNA-seq data 분석법 괸련 글을 토대로 KOBIC 교육영상 예제 데이터를 분석해보는 연습을 해보겠습니다.
[당신의 논문 동료] scRNA-seq data 분석법 – Plot 4 종류부터 DEG까지 | 뉴스 > Bio뉴스 > 동향 | BRIC
1. 들어가며 R을 활용하면서 가장 많이 나오는 에러에 대해 짚고 넘어가고 합니다. 1.1. R은 대소문자를 매우 엄격히 구분...
www.ibric.org
우선 전에 만들었던 'pbmc_tutorial1.rds' 데이터를 클릭을 통해 불러왔습니다.

다음으로 전에 설치했던 Seurat 패키지를 library로 다시 불러오고, DimPlot( ) 함수를 통해 다시 UMAP을 그려보겠습니다.
library(Seurat)
DimPlot(pbmc_tutorial1)

그럼 이제 DimPlot 함수를 세부 조정하여 다양한 형태의 UMAP을 그려보고자 합니다. 지금까지 그래왔듯 글쓴이께서 했던 부분들을 따라 해보며 연습해보려 하는데요, 우선 point size를 'pt.size' 옵션을 통해 조정해보고자 합니다.
DimPlot(pbmc_tutorial, pt.size = 2)
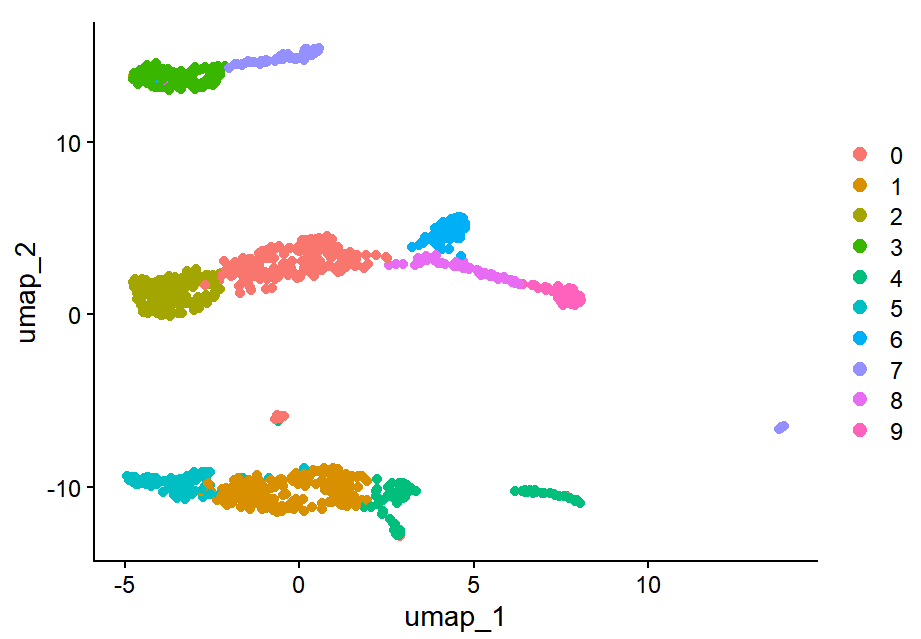
포인트가 확연히 커졌음을 확인할 수 있습니다. 참고로 방법은 같아도 활용한 데이터가 달라서 글에서의 UMAP 형태와 제 이미지의 UMAP 형태가 다릅니다. 이번엔 3번 클러스터만 초록색으로 하고, 나머지는 회색으로 해보겠습니다.
DimPlot(pbmc_tutorial1, cols = c("grey", "grey", "grey", "green", "grey", "grey", "grey", "grey", "grey","grey"))

다음으로 label을 표시해보도록 하겠습니다.
DimPlot(pbmc_tutorial1, label = T)

이제 X축과 Y축을 없애보고자 하는데요, 이땐 다음과 같이 NoAxes( )를 더해줍니다.
DimPlot(pbmc_tutorial1) + NoAxes()

그럼에도 아직 남아있는 범례(legend)가 있는데 이것도 NoLegend()를 추가적으로 더해 없애줄 수 있습니다.
DimPlot(pbmc_tutorial1) + NoAxes() + NoLegend()

이번엔 파악하고자 하는 유전자가 어디서, 얼마만큼 발현하는지 알 수 있는 함수를 활용해보고자 하는데요, UMAP 위에 유전자 발현량을 나타내어 주는 'FeaturePlot( )'입니다.
글에서 'SREBF1'을 파악하고자 하는 유전자로 활용한 만큼 저도 해당 유전자가 어떤 세포에서 얼마만큼 발현되는지 확인해보겠습니다.
FeaturePlot(pbmc_tutorial1, "SREBF1")

이게 많은건지 적은건지는 모르겠지만 Cluster 6에서 상대적으로 높게 발현하고 있는 듯 합니다.
잘 안보이니 점의 크기를 2배로 키우고, 뒤에 파묻힌 SREBF1 발현 세포들을 'order = T'라는 옵션을 통해 맨 앞으로 가져와보겠습니다.
FeaturePlot(pbmc_tutorial1, "SREBF1", pt.size= 2, order = T)

이렇게 확대해보니까 Cluster 6은 물론 Cluster 1과 2에서도 상대적으로 높게 발현되는 듯 합니다. 사실 뭔지 모르는 수준인지라 색깔이 있다 이 정도만 이해되는 것 같습니다.
이번엔 B cell marker인 'MS4A1'의 발현량을 확인해보겠습니다.
FeaturePlot(pbmc_tutorial1, "MS4A1")

와 이거는 상대적으로 높게 발현된다는 수준을 넘어 cluster 3와 7에서 높게 발현되고 있네요.
이번엔 두 유전자의 발현량을 함깨 조사해보고자 하는데, MS4A1과 유사하게 발현하는 것이 CD79A라 하는 만큼 이 두 유전자가 얼마나 같이(또는 따로) 발현고있는지 확인해보겠습니다.
FeaturePlot(pbmc_tutorial1, c("MS4A1", "CD79A"), blend = T)

이 또한 + NoAxes(), + NoLegend()를 이용해 X축, Y축, 범례를 없앨 수 있다고 합니다.
다음 글에서는 VlnPlot, DotPlot, 그리고 밝혀진 마커 유전자들을 토대로 cluster annotation 하는 방법을 정리해보도록 하겠습니다.
앞서 말했듯 제대로된 학습을 희망하시는 분들은 제 글을 보기보단 글 첫부분에 링크된 BRIC 글을 참고하시길 바랍니다. 감사합니다!
참고자료
1) INCOEDU, KOBIC 교육센터, 예제 데이터를 활용한 단일세포 전사체 데이터 분석
2) 쿼카, [당신의 논문 동료] scRNA-seq data 분석법 – Plot 4 종류부터 DEG까지, BRIC




