오늘 글부터는 Translation 관련 이론 복습을 해보도록 하겠습니다!
Translation은 간단히 말해서 mRNA에 아미노산이 붙은 tRNA가 적합한 mRNA의 codon에 맞게 붙으면서 아미노산들이 결합되어 protein이 합성되는 과정입니다. 본 단백질 합성 과정은 cytoplasm에 있는 rybosome에 의해 이루어집니다.
아래 영상에서는 Protein synthesis가 이루어지는 과정에 대해 재미있게 설명해주고 있습니다.
이러한 translation 과정(번역 과정)은 prokaryotes와 eukaryotes이 서로 비슷하며, initiation, elongation, 그리고 termination 단계로 이어집니다. 그렇게 20가지 종류의 아미노산들이 peptide 결합으로 연결이 되면서 단백질이 만들어지는 것이죠.
아미노산에 상응하는 codon을 알려주는 codon chart를 읽는 방법은 아래 영상을 참고하시면 되겠습니다.
진핵과 원핵 사이에 번역 과정이 비슷하긴 하나 initiation 과정은 차이는 있기에 먼저 원핵 세포(prokaryotes)에서의 translation에 대해 설명해보도록 하겠습니다.
Intiation Phase
영상으로 translation 과정을 만들어봤는데, 절차가 좀 길다보니 영상도 길어졌습니다. 그만큼 영상을 만드는데 시간도 많이 걸렸고, 그래서 기억에 오래남지 않을까 기대해봅니다(가볍게 만든 영상이 오류가 있을 수 있으니 양해부탁드립니다. 다른 영상을 보니 IF2가 aa-tRNA와 함께 P site에 붙는것 같기도 합니다).
글로 설명하자면, 우선, Bacteria mRNAs의 Shine-Dalgarno sequence(initiation codon 이전 5~10 nt)를 small ribosomal unit의 16S ribosomal RNA가 인식합니다.
이후 AUG sequence인 initiation codon에 small unit의 30S subunit이 상보 서열로 상호작용하게 됩니다.

위 그림을 보시면 small unit에 initaition factors(IFs)들이 붙어있는데, IF1은 mRNA에 대한 30S subunit(A site에 위치)의 attachment를 안정화하여 P site에 있는 initiator aa-tRNA가 A site로 가지 않도록 하고, IF3의 activity를 자극합니다 1).
IF3은 large subunit(50S)이 prematurely하게 붙는 것을 막아 initiator aa-tRNA가 적절하게 리보솜으로 진입할 수 있도록 돕습니다.
또한, IF3는 ribosomal P-site에서의 fMet-tRNAfMet의 결합을 안정화하고, mismatched codon-anticodon interaction을 불안정하게 하는 profreading capability도 갖는다1)고 합니다.
IF2는 initial aa-tRNA의 P site 부착에 필요한 GTP-biding protein인데, AUG codon에 해당하는 아미노산이 methionine을 가진 methionly-tRNAs이 AUG 코돈이 위치한 ribosome에서의 P site에 위치할 수 있도록 돕습니다.
이러한 과정을 통해 코돈이 잘못 해석되는 'frame shifting'* 현상을 막을 수 있는 것입니다.
Translational frameshifting : 리보솜의 원래 위치에서 한개의 뉴클레오티드만큼 옆으로 이동하여 새로운 단백질이 생성되는 과정4)
그렇게 initiation site에 붙고나면 IF1과 IF3가 방출되고, large subunit(50S)이 붙으면서 IF2에 결합한 GTP이 가수분해(hydrolyze)됩니다.

이러한 GTP hydrolysis는 리보솜의 형태를 변화시키면서 GTP는 GDP가 되고, IF2-GDP는 방출되며, 리보솜은 70S intiation complex가 되어 translation이 본격적으로 시작되게 됩니다.
여기서 GTP(guanosine triphosphate)는 transcription 단계에서도 등장했던 기억이 나지 않나요?
[대학원 준비 5일차] transcription in eukaryotes, 5' methyIguanosine cap(5' capping)과 3
이번엔 rRNA, tRNA에 이어 mRNA의 합성과 프로세스 과정에 대해 살펴보겠습니다. [대학원 준비 5일차] 이론 복습 :: U20 snoRNA, U68 snoRNA, 그리고 tRNA(transfer RNA)오늘은 4일 차 글에 이어 Transcription 관련 이
tkmstudy.tistory.com
GTP는 RNA 합성에 필요한 building block이면서도 GTPases에 의해 GTP가 GDP (guanosione diphosphate)로 hydrolysis되는 과정에서 energy가 방출되기에 protein synthesis 과정에서 energy source로 사용된다2)고 합니다.
여기까지가 translation의 initiation 단계이고 elongation 단계로 넘어가겠습니다.
Elongation Phase
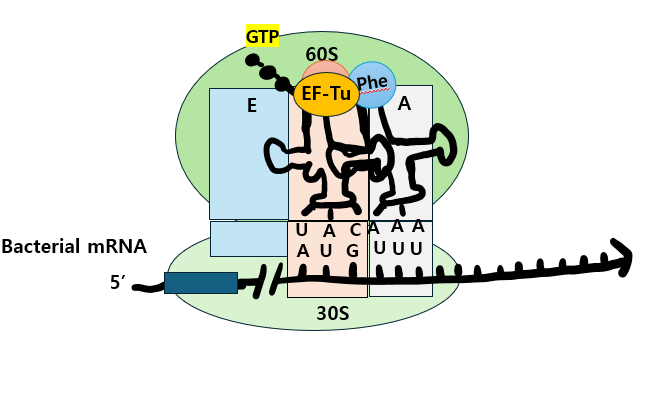
본 단계에서는 먼저 Methyl-tRNA가 P site에 자리잡게 된 이후 EF(elongation factor)-Tu 혹은 Tu라 불리는 GTPase가 두번째 aminoacyl-tRNA가 A site로 가도록 돕습니다.

참고로, EF-Tu는 bacteria에서 highly conserved 되어있을 뿐만 아니라 가장 풍부하게 존재하는 단백질3)이라고 합니다.
그렇게 correct-aminoacyl-tRNA Tu-GTP complex가 만들어지고 나면 리보솜에서 conformational change가 일어나 aa-tRNA가 P site에서 mRNA에 bound된 상태로 유지되게 됩니다. 그 과정에서 GTP는 GDP로 가수분해되면서 Tu-GDP complex가 방출이됩니다.
이후에는 두개의 amino actides(aa) 사이(사진에선 Met와 Phe)에서 peptide bond가 형성되는데, 구체적으로 A site의 aa-tRNA의 amine nitrogen이 P site의 tRNA에 결합한 아미노산의 carbonyl carbon에 nucleophilic attack을 하게됩니다.
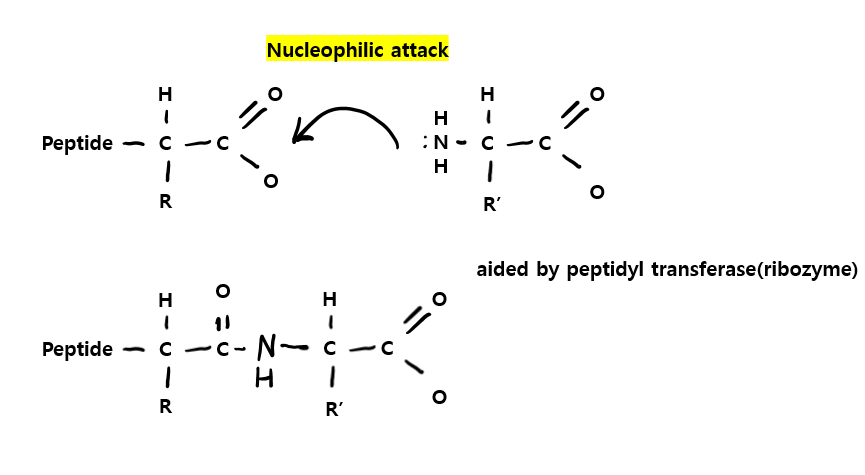
그렇게 되면 external energy의 필요 없이 peptide bond가 형성됩니다. 이 과정은 ribsome의 large subunit의 구성요소인 peptidyl transferase(ribozyme)에 의해 촉매됩니다.
결과적으로 A site의 tRNA는 dipeptide가 되고, P site의 tRNA는 deacylated되게 되며, mRNA의 5'에서 3'으로 리보솜이 이동하게 됩니다. 이 단계를 'Translocation 단계'라고 지칭합니다.
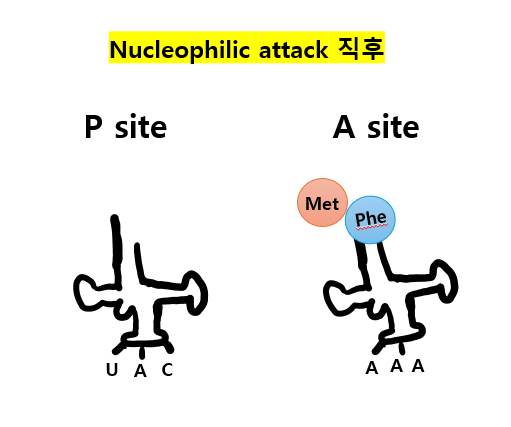
이후 리보솜이 mRNA의 5' -> 3' 방향으로 이동하며 dipeptdyl-tRNA가 A site에서 P site로 이동하고, deacylated tRNA에는 P에서 E site로 이동하는 elongation 과정이 이어지게 됩니다.
이때 tRNA의 anticodons와 mRNA의 codons가 hydrogen bond를 잘 유지할 수 있는 건 EF-G라는 GTP-bound elongation factor가 ribosome을 안정화시켜주기 때문입니다.
EF-G의 역할이 EF-Tu와 역할이 비슷해보이는데, EF-Tu-GTP는 aminoacyl-tRNA를 선택할 때, EF-G-GTP는 translocation 과정에서 리보솜을 안정화해준다고 이해하면 될 것 같습니다. 결국, 각 cycle마다 2개의 GTP가 사용됩니다.
참고로 선행 논문5)에 따르면, EF-G에 의한 GTP hydrolysis의 주요 역할은 tRNA나 mRNA의 movement가 아니라고 주장하는데
EF-G가 단순히 tRNAs를 ribosome을 통해 밀치는 것(push)이 아니라(A-> P & P -> E), tRNAs의 움직임을 제어(restrain)하는 것이라고 합니다(translocation step이 정확하게 이루어지도록).
그렇게 EF-G의 GTP hydrolysis는 리보솜의 형태적 변화를 일으키면서 A site는 텅 비게되고, 이 과정에서 EF-G-GDP는 리보솜으로부터 dissociate됩니다.
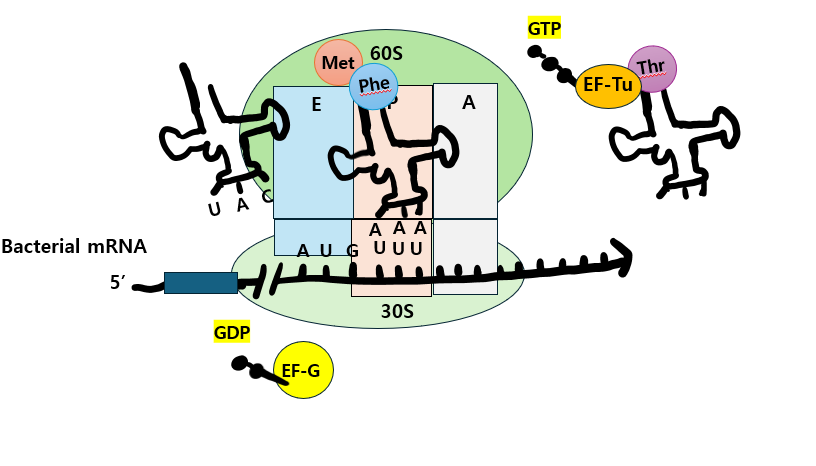
이후 E site에 있는 deacylated tRNA는 리보솜을 떠나게 되고, A site로는 새로운 aa-tRNA가 들어오게 되고, 이러한 elongation cycle이 stop codon을 만나기 전까지 계속 이어지게 됩니다.
아래 영상으로 보면 더 직관적으로 잘 이해되실 수 있지 않을까 싶습니다.
다음으로 Eukaryotes에서의 translation 과정을 설명해보도록 하겠습니다. 사실 initiation 부분만 특이적으로 달라서 translation을 위한 intiation 단계에 대해서만 설명해보겠습니다.
우선 eukaryotic ribosomes는 60S large subunit과 43S small subunit을 갖는데, bacterial ribosomes보다 큽니다.
무엇보다 최소 12개의 intiation factors를 필요로 하며, 43S complex의 40S subunit에는 여러 eIFs(eIF1, eIF1A, eIF3, eIF5)와 eIF-GTP를 가진 initiator tRNA가 결합하게 되며 이것이 mRNA에 결합하기 위한 준비과정이라고 볼 수 있습니다.
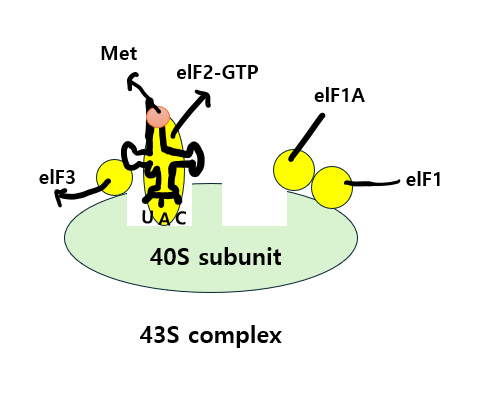
단계적으로 설명하자면, eIF4E가 mRNA의 5' cap에 결합하고, eIF4A는 mRNA의 5' end를 따라 이동하면서 43S complex의 움직임을 방해할 수 있는 double strand regions를 제거합니다.
이후 eIF4G는 5' capped end와 mRNA의 3' poly(A) 사이에 linker로서 역할을 하며, liner mRNA를 circular로 바꿉니다.
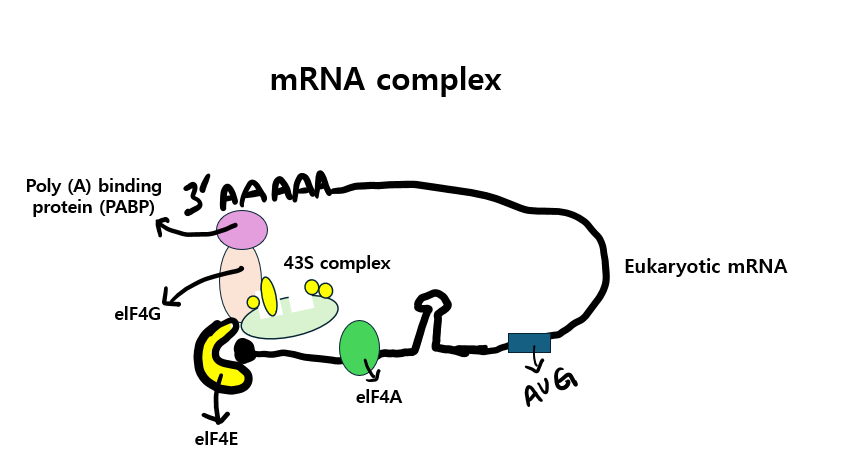
이후 43S preinitiation complex가 형성되고 나면, 5' mRNA를 찾을 수 있게 되는데 이때 eIF3과 eIF4G 사이의 protein-protien interactions가 유도되게 됩니다. 그렇게 small subunit(43S complex)이 strand를 따라 이동하다가 적절한 AUG codon을 찾았다면 eIF2-GTP는 eIF3-GDP로 hydrolyze되면서 방출되고, large subunit (60S)이 붙으며 initaition phase를 마치게 됩니다.
참고로 AUG codon은 Kozak sequence (5' CCACCAUGC 3')에 포함되며, complete 80S ribosome이 형성되기 위해선 또 다른 GTP-binding protein인 elF5B-GTP가 가수분해되어야 한다고 합니다. 즉, 진핵 세포의 initiaion 단계에서는 translocation 단계에 가기 전 두 개의 GTP의 hydrolysis를 필요로 하는 것입니다.
위 영상은 앞서 prokaryotes의 translation의 개시를 돕는 sequence인 bacterial mRNAs의 'Shine-Dalgarno sequence'와 eukaryotes의 translation의 개시를 돕는 sequence인 eukaryotic mRNAs의 'Kozak sequence'에 대해 설명해주고 있으니 참고바랍니다.
전반적인 eukaryotic cells에서의 translation 절차는 아래 영상을 참고하시면 되겠습니다.
마지막으로 Termination(종결)이 어떻게 이루어지는지 설명해보도록 하겠습니다.
Termination Phase
Termination은 UAA, UAG, UGA라는 세 가지 stop codon을 만나게 되면 이루어지는데, 그러려면 release factors(RFs)가 필요합니다.
이때 RFs가 A site로 들어가면 mRNA의 several nucleotides에 영향을 주는 리보솜 small subunit의 conformational change를 유발하고,
인접한 polypetide와 tRNA 사이를 linking하는 ester bond가 hydrolyze되면서 completed polypeptide가 방출됩니다.
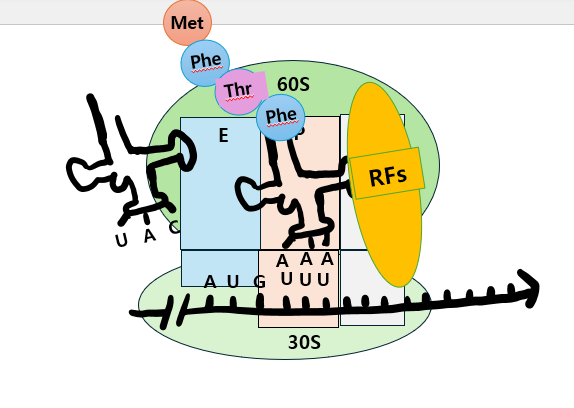
즉, translation의 마지막 단계는 P site에 있는 decylated tRNA를 방출시키고, 리보솜으로부터 mRNA를 dissociate시키고, 다음 번역을 준비하기 위해 ribosome을 large & small subunit으로 분해합니다.
참고로 stop codon과 관련해서 'nonsense mutation'이라는 현상이 있습니다.
이 돌연변이는 gene의 coding sequence 내에서 비정상적으로 stop codons이 생기게 하는 것으로, 사람의 유전 장애 중 대략 30%를 차지할 정도라고 하네요.
이러한 nonsense mutation은 하나의 nucleotide substitutions를 변화시켜서 아미노산을 coding하던 sense codon을 nonsense 혹은 premature termination codon(PTC)로 변형시키게 되고,
PTC-contaning mRNA는 nonsense-mediated mRNA decay(NMD)라 불리는 surveillance pathway로 분해되거나 non-functional한 shortened protein을 생산하게 될 수 있다6)고 합니다. 무엇보다 이러한 과정은 human diseases의 병리학적 기전으로 이어질 수 있기에 nonsense mutation을 억제하는 치료 전략을 세우기 위한 연구가 진행되고 있는 중이라고 합니다.
참고로 본 돌연변이는 부정확하거나 비효율적인 pre-mRNA splicing 혹은 부적절한 RNA editing에 의해 나타날 수 있다7)고 합니다. mRNA-splicing에 대한 설명은 아래 글을 참고하시면 되겠습니다.
[대학원 준비 6일차] mRNA splicing, Exonic splicing enhancers(ESEs), 그리고 self-splicing
오늘은 전에 5' capping과 3' tailing으로 만들어진 pre-mRNAs가 mature mRNAs가 되기 위해 필요한 RNA splicing 절차에 대해 소개해보도록 하겠습니다. [대학원 준비 5일차] transcription in eukaryotes, 5' methyIguano
tkmstudy.tistory.com
지금까지 eukarytoes와 prokaryotes에서의 단백질 합성 과정인 translation 과정을 정리해보았습니다.
여기서 중요한건 단백질 합성 과정은 리보솜 하나에서만 일어나는 것이 아니라 리보솜 여러 개가 하나의 mRNA에 polyribosme이라는 complex 형태로 붙어 동시다발적으로 진행하게 된다는 것입니다. 아래 영상에서처럼 말이죠.
즉, 각각의 리보솜들이 5' cap에서 small subunit으로 붙어 AUG(initiation codon)를 인식하면 complete complex를 이루어 translation을 시작하면서 이동을 하는데,
하나의 ribosome이 initiation codon을 떠나게 되면, 다른 리보솜이 거기에 붙어 translation activity를 갖게 되고, 그렇게 여러 개의 리보솜이 하나의 mRNA에 붙어서 한번에 많은 단백질들을 동시에 합성할 수 있게 되어 protein sythesis의 속도가 빨라지게 됩니다.
translation을 하나의 글에 정리하려다 보니 내용이 길어지게 되었네요.
이론 베이스를 확실하게 잡아두기 위해 당분간은 이론만 복습해보도록 할 예정인지라 다음 글은 gene expression 제어와 관련한 이론을 복습해보도록 하겠습니다. 감사합니다!
참고자료
1) Laursen BS, Sørensen HP, Mortensen KK, Sperling-Petersen HU. Initiation of protein synthesis in bacteria. Microbiol Mol Biol Rev. 69(1):101-23 (2005).
2) HMDB, Showing metabocard for Guanosine triphosphate (HMDB0001273), 2021, URL : https://hmdb.ca/metabolites/ HMDB0001273
3) Inter Pro, Elongation factor Tu (EF-Tu), GTP-binding domain > Description, URL : https://www.ebi.ac.uk/interpro/ entry/InterPro/IPR041709/
4) NAVER 지식백과, 번역틀변동(translation frameshifting)
5) Rexroad G, Donohue JP, Lancaster L, Noller HF. The role of GTP hydrolysis by EF-G in ribosomal translocation. Proc Natl Acad Sci U S A. 119(44):e2212502119 (2022).
6) Morais P, Adachi H, Yu YT. Suppression of Nonsense Mutations by New Emerging Technologies. Int J Mol Sci. ;21(12):4394. (2020).
7) Keeling KM, Du M, Bedwell DM. Therapies of Nonsense-Associated Diseases. In: Madame Curie Bioscience Database [Internet]. Austin (TX): Landes Bioscience; 2000-2013. Available from: https://www.ncbi.nlm.nih.gov/books/NBK6183/




