Bacterial Operon에 이어 이번엔 Eukaryotes의 gene expression 조절 기전에 대해 살펴보도록 하겠습니다.
[10일차] Lac Operon(structural genes, promoter, operator), 그리고 lactose와 관련된 β-galactosidase 등의 효소
오늘은 Translation 관련 이론 복습에 이어 Gene Regulation 관련 이론 복습을 해보도록 하겠습니다. [9일차] Translation 과정 :: Initiation, Elongation, Termination 그리고 Nonsense Mutation오늘 글부터는 Translation
tkmstudy.tistory.com
오늘 논문을 읽다가 gene expression에 대한 인상 깊은 문장을 발견해서 인용하며 시작하겠습니다.
"Differential gene expression mechanisms ensure cellular differentiation and plasticity to shape ontogenetic and phylogenetic diversity of cell types",
Anil Panigrahi & Bert W. O’Malley0).
(서로 다른 gene expression 메커니즘은 세포 간의 차이와 세포 유형의 다양성을 가능하게 했다는 뜻)
Eukaryotes의 Gene expression은 4가지 level로 구분할 수 있으며 각각 Transcriptional control, Processing control, Translational control, Posttranslational control입니다. 하나씩 정리해보도록 하겠습니다.
1. Transcriptional Control (전사 제어)
우선, 전사 과정에는 Transcriptional Factors(TFs)가 주요 역할을 합니다. TFs에 대한 설명은 아래 글을 참고하시면 되겠습니다.
[대학원 준비 5일차] transcription in eukaryotes, 5' methyIguanosine cap(5' capping)과 3
이번엔 rRNA, tRNA에 이어 mRNA의 합성과 프로세스 과정에 대해 살펴보겠습니다. [대학원 준비 5일차] 이론 복습 :: U20 snoRNA, U68 snoRNA, 그리고 tRNA(transfer RNA)오늘은 4일 차 글에 이어 Transcription 관련 이
tkmstudy.tistory.com
윗 글에 소개한 TFs 말고도 정말 많은 TFs가 genome 곳곳에서 유전자 발현을 control하는데요, 특정 유전자의 다양한 regulatory sites에 결합하는 sequence-specific transcription factors는 transcription activators와 transcription inhibitors로 구분할 수 있습니다.
지금까지 sequence-specific TFs들이 regulatory programs를 조율한다는 것은 밝혀지긴 했지만, 많은 regulatory elements가 유사한 TFs 그룹이 타깃하는 sequence motifs를 가지고 있는 것은 물론, 대부분의 TFs가 regulatory sequences에 wide-spread binding을 보여주고 있어 DNA sequence 하나로 gene expression patterns를 예측하는 것은 거의 불가능하기에 각 TF의 multiple regulatory mehanisms의 combined impact를 모델링하고 검증하기 위한 연구가 진행되고 있다1)고 합니다.
재밌는 건 이러한 transcription factors를 embryonic stem cells(ES cells)*에 활용하면, 해당 세포들을 원하는 세포 유형으로 reprogram할 수 있다는 것입니다.
embryonic stem cell (배아줄기세포) : 어떤 유형의 세포나 조직으로든 분화될 수 있는 pluripotency를 가진 세포 2)
그러나 이러한 ES cells는 윤리적인 문제가 있죠. 그에 대한 대안으로 2006년 야마나카 신야 교수의 발견으로 ES cells처럼 역할을 하는 iPS(induced Pluripotent Stem) cells가 개발되었습니다.
iPS cells가 무엇이냐 하면 4가지의 특정 transcription factors (Oct4, Sox2, c-Myc, and Klf4)를 인코딩하는 genes의 조합을 adult fibroblast cells에 주입하여
fibroblasts를 리프로그래밍함으로써 이들을 ES cells처럼 행동하는 undifferentiated cells로 전환하는 것입니다.

이와 같이 체세포에 줄기세포 능력을 부여하는 생체 리프로그래밍 방식은 인체 장기의 재생을 촉진시키는 것은 물론, 노화 특성을 역전 3)시킬 수 있어 학계와 산업계에 큰 주목을 받았죠.
무엇보다 TF가 DNA sequence에 결합하면 본 서열이 nucleases에 의해 digestion되지 않게 보호되는데요, 이 특징을 활용해 Transcription에 관여하는 genome의 서열을 분석하는 Genome-Wide Association Study(GWAS)의 하나인 ChIP(Chromatin immunoprecipiation)을 진행할 수 있습니다.
아래 영상을 보시면 ChIP으로 어떻게 DNA 서열을 분석하는지 이해가 쉽게 되실겁니다.
간단히 글로 설명하자면, ChIP-seq는 TF에 결합한 DNA 서열을 identification하는 방법으로 우선 Formaldehyde를 사용하여 DNA에 proteins가 붙은 상태로 유지되도록 고정하고, 특정 효소를 사용해 small DNA fragments로 만듭니다.

이후 특정 proteins(TF)에 결합하는 항체(antibody)를 사용하여 immunoprecipitation(침전)시키고, magnetic beads 등으로 purification을 시켜 DNA에 bound된 antibody에 결합된 특정 proteins(TFs)를 뽑아냅니다.
그리고나서 heat incubation 혹은 protein component의 digestion을 통해 crosslinking을 분해시키고, 뽑아낸 DNA sequence에 대해 서열분석을 진행합니다. 그러면 특정 TF가 어떤 genes를 활성화하는지 알 수 있게 됩니다.
다음으로 transcriptional activators에 의한 'Transcriptional Activation(TA)'에 대해 알아보도록 하겠습니다. 우선 TA에 관여하는 요인으로는 Enhancers, Promoters, 그리고 Coactivators가 있습니다.
먼저 Enhancers는 sequence-specific transcriptional activators를 위한 multiple binding sites를 포함하는 DNA elements로
looping을 통해 target promoter와 distant communication이 가능하며 이들은 promoter와 enhancer 각각에 결합된 proteins를 통해 상호작용을 한다4)고 합니다.
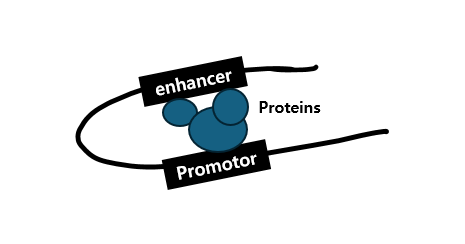
그러면서도 선행 논문5)에 따르면, enhancer에 의한 transcription level은 promoter로부터의 genomic distance(P-E)와 contact probability를 내포한 non-linear relationship에 의해 결정된다고 합니다.
Enhancer가 무엇이며 Enhancer를 검출하기 위해 어떤 방식이 활용될 수 있는지 아래 영상에서 설명해주고 있으니 참고바랍니다.
참고로 promoter (translation initiation site recognized by TFs)와 enhancers는 'insulators'라는 또다른 DNA sequence elements에 의해 "cordoned off"가 될 수 있습니다.
왜이리 귀찮게 transcription을 조절하는 장치를 복잡하게 만들었나 싶을 수 있지만, transcription과 translation 모두 우리의 몸을 구성하고 기능하게 하는 중심 원리이기에, 우리 몸에서 이러한 과정이 부적절하게 이루어지면 우리의 생존이 위협받을 수 있습니다.
몸이라는 시스템이 지속되는데 결국에 가장 중요한 건 '생존'일 것이고, 그래서 본 주요(=고위험) 과정들이 특수하게 관리될 수 있도록 진화적으로 복잡하고 어쩌면 귀찮게 설계된 것이 아닐까 싶습니다.
사족이 길어졌는데 그렇게 insulator는 genome의 adjacent regions 사이의 부적절한 interactions이 발생하는 것을 막는다6)고 합니다.
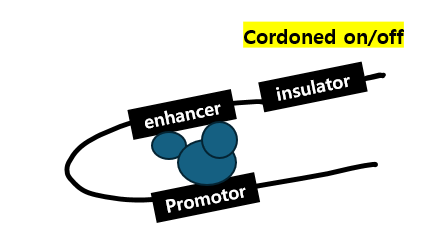
만약 insulator가 스위치를 키면(cordoned on) enhancer에 결합한 transcription activator는 coactivator를 통해 transcription 과정을 자극합니다.
이 co-activators는 catalytic multiprotein complex로서 transcription에 필요한 DNA anchored TFs와 상호작용할 수 있는 잠재력이 있을 뿐 아니라 효과적인 transcription이 이루어질 수 있도록 acetylation, demethylation과 같은 특정 epigenetic modifications를 regulate한다7)고 합니다.
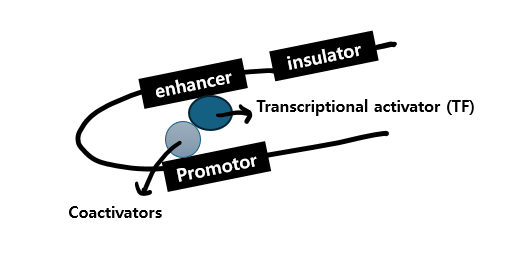
그렇게 co-activators는 transcriptionary machinery(TFs, RNA polymerase II)를 이용 불가능한 상태에서 이용 가능한 상태로 바꿉니다.
참고로 enhancer에 결합한 TFs와 basal transcription machinery 사이에 소통하는 coactivator로 'mediator'라는게 있는데 이것은 RNA polymerase II와 직접적으로 상호작용 합니다. 아래 이미지를 보시면 이해가 쉽게 되시지 않을까 싶습니다.
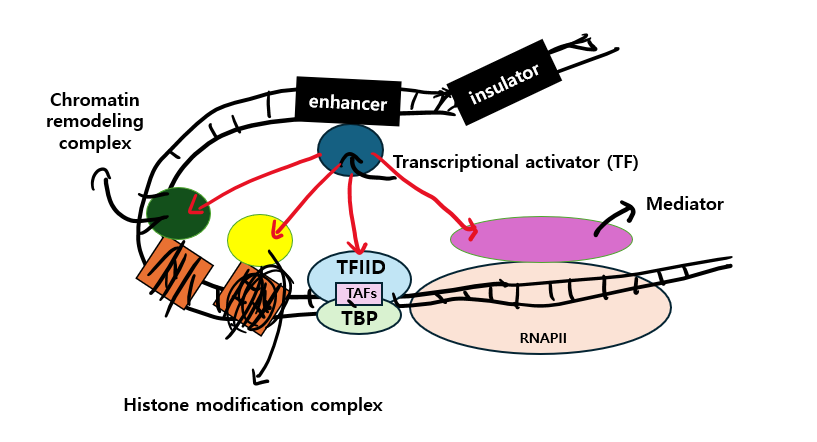
또한, 앞서 말했듯 coactivator는 epigenetic modifications를 수행하는데 chromatin의 histones를 변형함으로써 chromatin의 구조를 변화시켜 histone acetyltransferases(HATs)를 통해 Acetyl Groups를 core histones의 lysine residues에 추가할 수 있습니다.
본 역할을 수행하는 Chromatin remodeling complex는 ATP hydrolysis를 통해 에너지를 사용하고, nucleosome structure를 변화시켜 DNA의 regulatory sites에 다양한 단백질들이 결합될 수 있도록 합니다.
아세틸화 과정에 대해 deacteylated histones로 엮인 DNA의 GRE(glucocorticoid response element)* 영역에 GR(glucocorticoid receptor)이 오게 된 상황으로 설명해보겠습니다.
* GRE: palindrome DNA sequence (5'-AGAACA-nnn-TGTTCT-3')
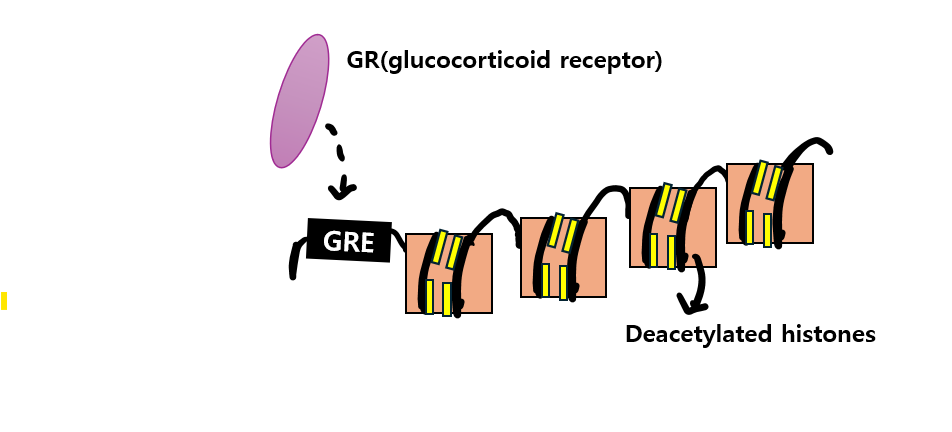
우선 GR에 GRE와 HAT units를 포함한 CBP(Histone Acetyltranferase)가 결합하고 나면, CBP는 histones의 특정 lysine residues의 amino groups에 acetyl groups를 transfer하고, TATA box로부터의 upstream와 downstream의 histones가 acetylated됩니다.
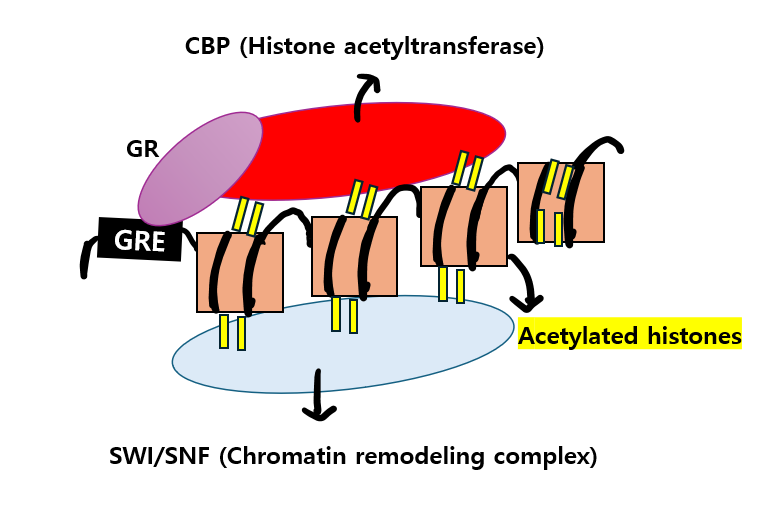
Acetylated histones에는 chromatin remodeling complex인 SWI/SNF가 recruit되고, CBP와 SWI/SNF는 함께 chromatin의 구조를 더 open시킵니다.
다음에 TFIID가 DNA의 open region(TATA box)에 결합하고 나면, TFIID의 subunit인 TAFII250 또한 histone acetyltransferase activity를 갖게됩니다. 그렇게 CBP와 TAFII 250은 함께 또 nucleosomes을 변화시켜 transcription initation이 가능하도록 합니다.
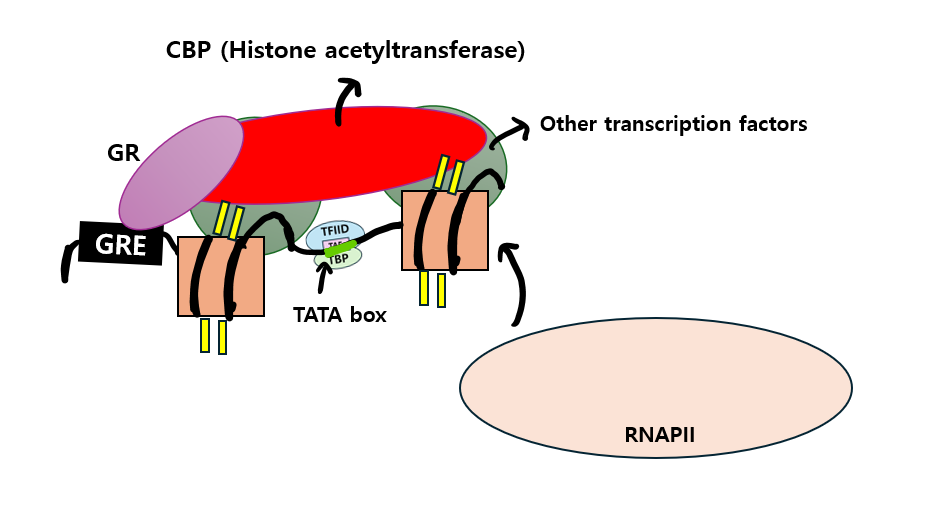
그렇게 acetylated된 상태에서 promoter에 RNA polymerase II가 결합하게 되고, transcription이 시작하게 되는 것입니다.
글이 길어진 관계로 transcription activation을 억제하는 즉, transcription repression을 담당하는 DNA methylation에 대해선 다음 글에서 소개하도록 하겠습니다.
참고자료
0) Panigrahi, A., O’Malley, B.W. Mechanisms of enhancer action: the known and the unknown. Genome Biol 22, 108 (2021).
1) Duttke, S.H., Guzman, C., Chang, M. et al. Position-dependent function of human sequence-specific transcription factors. Nature 631, 891–898 (2024).
2) NAVER 지식백과, 생화학백과, 배아줄기세포
배아줄기세포
배아줄기세포(embryonic stem cell)는 배반포의 내부 세포 덩어리로부터 분리해 낸 줄기 세포로 세포 증식이 가능하고 우리 몸에 필요한 어떤 종류의 세포나 조직으로도 분화할 수 있는 능력인 전분
terms.naver.com
3) Bio통신원, 생체 리프로그래밍으로 ‘재생 줄기세포’ 생성과 소장 재생 기전 밝혀, BRIC, 2023, URL: https://www.ibric.org/bric/ trend/bio-news.do?mode=view&articleNo=9875604#!/list
4) Kulaeva OI, Nizovtseva EV, Polikanov YS, Ulianov SV, Studitsky VM. Distant activation of transcription: mechanisms of enhancer action. Mol Cell Biol. 32(24):4892-7. (2012).
5) Zuin, J., Roth, G., Zhan, Y. et al. Nonlinear control of transcription through enhancer–promoter interactions. Nature 604, 571–577 (2022).
6) Gaszner, M., Felsenfeld, G. Insulators: exploiting transcriptional and epigenetic mechanisms. Nat Rev Genet 7, 703–713 (2006).
7) Talukdar, P.D., Chatterji, U. Transcriptional co-activators: emerging roles in signaling pathways and potential therapeutic targets for diseases. Sig Transduct Target Ther 8, 427 (2023).




